化合物-タンパク相互作用 (chemical-protein interaction, CPI) 予測モデルを開発するには、化合物とタンパクを数値に変換することがまず必要です。ここでは代表的な手法について紹介します。
低分子化合物の表現方法
化合物は,文字列やグラフ,画像など,さまざまな形式で記述することができます。その中で最も広く使われているのは文字列形式で、特にSimplified Molecular-Input Line-Entry System (SMILES) が古くから使われてきました (J Chem Inform Comput Sci. 1988)。SMILESは、化合物を線状の文字列として記述する方法です。1つの原子から始まり、閉じた環系の結合をたどってすべての原子を訪問します。また、原子、結合、環状構造、分岐、立体化学それぞれに特化したルールで表現します。1つの化合物が複数のSMILESで表すことができますが、それを一意にするアルゴリズムに依存していくつかの正規化スキームがあります。例えば,ケモインフォマティクス (化学情報学) で最もよく使われているPythonライブラリであるRDKitは,SMILESを生成する際に分子の立体化学と対称性の両方を考慮するアルゴリズムを実装しています (J Chem Inform Modeling 2015)。また,SMILESの非射影性を補い,より偏りの少ない情報を生成するために,SMILESの拡張を利用する機械学習ツールもあります (arXiv 2017)。また,SMARTS (局所的な部分構造に着目する) やSELFIES (有効な化合物のみを生成できる)といった、SMILES以外の文字列表現も登場してきました (arXiv 2019) 。
いずれにせよこれらは文字列ですが、機械学習モデル構築の上では何らかの数値化をする必要があります。SMILESはワンホットベクトルとマルチホットベクトルの混合として符号化することができます。例えば、Hiroharaらは価電子数(VE)を正規化し、キラリティ、芳香族性などの化学構造をワンホットエンコーディングで原子値を割り当てて符号化しました (BMC Bioinform. 2018)。この方式は、SMILESを計算可能な形式に効果的に符号化するものです。多くの報告において、符号化されたSMILESのベクトルはさらに潜在的なベクトルを得るために深層学習モデルに供給されていきます (Nature Commun 2020)。SMILESを符号化するワンホットエンコーディングとは別の方法としてWord2vecもあり、これは固定長の文字を「単語」として扱い、化学構造全体の強力な埋め込みを生成することができます (Front Chem. 2020)。
化学物質に含まれる構成的な部分構造/足場や共通の官能基をもとにして、化学物質をその部分構造があるかないかというブール表現として記述するフィンガープリントを使うということも可能です。ECFP、Morgan、PubChem、MACCS などの異なるフィンガープリントがこれまで提案されてきました。大別すると、化合物のトポロジーに基づくフィンガープリント(Morgan、ECFP、2D pharmacophoreなど)とSMARTSに基づくフィンガープリント(MACCS、PubChemなど)に分類することができます。前者であるトポロジーベースのフィンガープリントは、分子内のトポロジカル距離を計算することによって原子と結合を特徴付け、SMARTSベースのフィンガープリントは、結合順序と結合芳香族性を記述するSMARTSパターンの存在を考慮したものです。
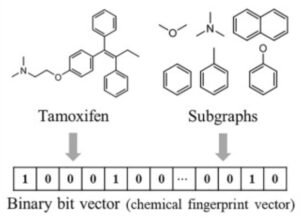
これらのフィンガープリントは直感的で情報量が多いため、フィンガープリントに基づく方法はケモインフォマティクスで古くから利用されてきました (Bioinformatics 2008)。また、2つの化合物の類似性はタニモト係数 (J Cheminformatics 2015)という対応するフィンガープリントベクトルを比較することによって簡単に計算することができます。
最近はこれらに加えてグラフニューラルフィンガープリントのようなグラフベースの表現が化学的特性を反映するのに成功しています (Chem Sci. 2018)。グラフベースの学習ストラテジーを利用するためには、化合物をグラフに変換する必要があり、典型的には原子や結合情報を持つグラフの隣接行列表現が使われています。

これらの行列をグラフ畳み込みネットワーク(GCN)の入力として使います。GCN の主な役割は、隣接するノード (頂点のこと) を考慮してノードの情報をアップデートすることです。この更新にも大きく2種類のタイプが有り、spectral GCN はグラフを全体として考慮し、spatial GCN はグラフの局所的な情報を考慮するものです。より一般的に使われているのはspatial GCNで、化合物のグラフ符号化に関する詳細は最近のレビュー論文 (Nat Rev Drug Discovery 2019)に記載されています。
ターゲットタンパク質の表現方法
タンパク質は基本的にアミノ酸配列であり進化的に高度に保存された情報です。このアミノ酸配列をone-hot法、word2vec法、k-mer法などで符号化することができます。このうち、one-hot符号化とは文字を2値ビットベクトルに変換するもの (BMC Bioinform. 2020)で、一例を次に示します。
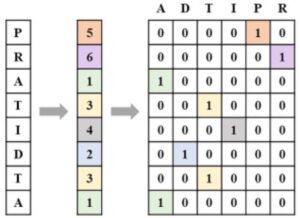
タンパク質構造をグラフとして表現する場合には、グラフのノードをアミノ酸残基とし、構造において2つの残基間が予め設定した距離内にあるときにエッジを結ぶという変換することが一般的です (spatially interacting graph)。

タンパク質の構造情報には、アミノ酸レベルの座標や静電特性、表面積が使われています (arXiv 2019; Cell Systems 2020; J Chem Inf Model 2019)。UniProtとProtein Data Bank (PDB)は、タンパク質の配列と構造情報に関する主要なリソースです。さらに、PDBにはリガンド特異的な空間コンフォメーションを含む化合物-タンパク質相互作用情報が含まれています。こうしたデータベースをマイニングすることで、タンパクの符号化に必要な情報を集めるわけです。
PDBの問題点は、構造的が解かれているタンパク質の数が、アミノ酸配列が分かっているタンパク質に比べてはるかに少ないことです。そのため、現状ではタンパク質の構造情報をコンピュータ支援薬物設計(computer-aided drug design, CADD)に利用することは限られていました。しかし、AlphaFold2の登場によりアミノ酸配列からタンパク質構造を予測できるようになりました。今後はAlphaFoldのデータベース情報を活用したタンパクの符号化も行われるようになるでしょう。
まとめに代えて
この記事では、AI創薬を行う上で、化合物とタンパクをどのように符号化するのかという点について概説しました。
創薬科学ははじめてだといろいろ難解な用語が出てきますが、それらを平易に解説した入門書がこちらの本です。
また、AIで予測した化合物が本当に薬として機能するかは、タンパクと化合物の相互作用を調べる必要があります。そういった実験手法をまとめたのがこちらの本です。
AI創薬研究については、このような関連記事があるので合わせてご覧ください。
今日も【生命医学をハックする】をお読みいただきありがとうございました。当サイトの記事をもとに加筆した月2回のニュースレターも好評配信中ですので、よろしければこちらも合わせてどうぞ