遠いホモログ配列を感度良く検出する方法がPSI-BLASTです。
この記事では、PSI-BLASTの基盤となっているPSSMやProfileの計算法について簡単に紹介した後、実際にPSI-BLASTを使っていきます。
位置特異的行列とProfile
多重配列アラインメント結果を目視すれば、どの位置に何が保存されているのかを知ることができますが、これを数値化したのが位置特異的行列やProfileです。
核酸でもアミノ酸でも原則は同じですが、アミノ酸よりも核酸の方が種類が少なく説明上好都合なので、DNA配列の場合を見ていきます。
とりあえず5つの配列があり、ギャップを考慮しないアラインメントがこのようになったとします。
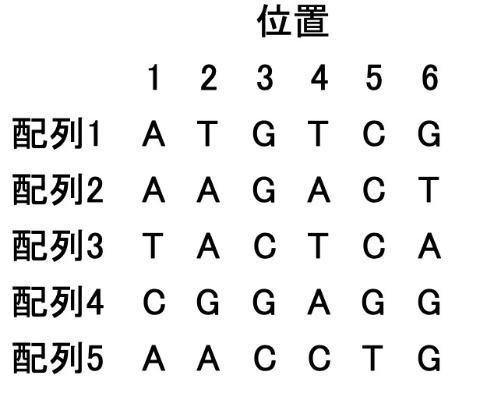
次に、それぞれの位置における頻度を計算します。例えば位置1においては5つの配列のうち3つがAなので、この位置におけるAの頻度は0.6になります。同様にして、全ての位置における頻度と、配列全体でのそれぞれの頻度を計算するとこのようになります。

そして、この「全体」の値で、それぞれの位置の値を割ります。例えば位置1のAにある0.6を、「全体」の0.3で割ると2.0になります。このような計算を行うと、このようになります。

この値のlog2を計算するとこのようになります。

この行列のことを、位置特異的スコア行列 (Position-Specific Scoring Matrix, PSSM) といいます。その多重配列アラインメントにおいて、それぞれの位置の塩基がどの程度保存されているのかを定量化した行列になっていて、値が高いほど保存性が高いということを意味しています。
例えば位置1や2においては、Aがよく保存されているということが分かります。
また、ある配列がこの多重配列アラインメント結果にどの程度当てはまるか、スコアとして計算することもできます。例えばAAGTCGという配列であれば、それぞれの位置における各塩基のスコアを加算していくことで

1.00 + 1.00 + 0.80 + 1.00 + 1.38 + 1.15 = 6.33と計算できます。
もともとこの行列は2を底とする対数で表示されているので、このスコア6.33が意味しているのは、ランダムな6塩基の配列と比較してAAGTCGは2の6.33乗、つまりおよそ80倍ほどこの多重配列アラインメントにマッチしているということです。
ここで紹介したのはギャップ (挿入や欠失) を考慮しないアラインメント結果からのスコア行列でしたが、ギャップを考慮したアラインメントも可能であり、同様にして計算した行列をProfileと呼びます。
このProfileを使って遠いホモログ遺伝子を次々に同定していく手法の1つがPSI-BLASTです。
PSI-BLASTとは
PSI-BLASTの概略を簡単に説明します。
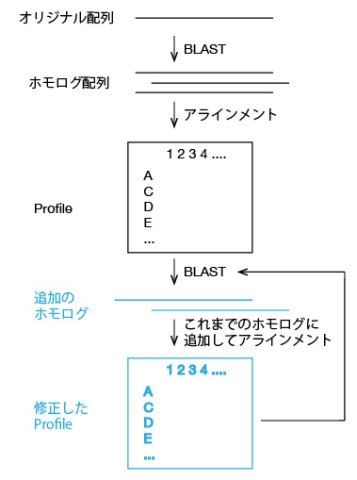
まずはある配列をもとにBLASTを行い、類似性の高い配列をまとめてアラインメントを作成します。
そのアラインメントを使ってProfileを作成し、次にそのProfile行列を使ってBLASTを再度実行することで、新しい配列を見つけることができます。
追加の配列も加えてProfileを作成し、再度BLASTをかけて、というのを繰り返すことで、ホモログ配列を検索するという方法です。
PSI-BLASTの使い方
PSI-BLASTは、NCBIが提供するBLASTのオプションを設定することで簡単に実行できます。
Protein BLASTのページにアクセスします。
今回はNCBI Protein データベースから古細菌のトランスポザーゼ の配列を使ってみます。FASTA形式のアミノ酸配列をコピーしてBLASTに貼り付けます。
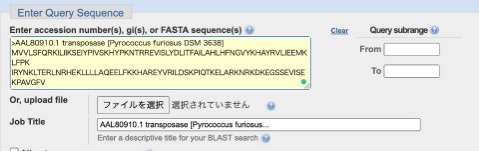
データベースは、「Reference proteins」を選択しておきます。
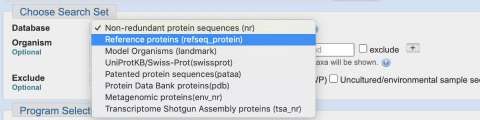
下の方にスクロールすると、Program Selectionという項目があり、ここでPSI-BLASTを選択できるようになっています。

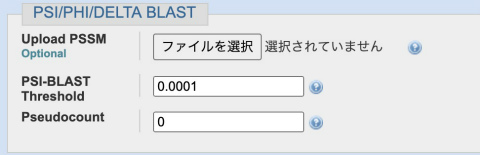
さらに、BLASTボタンの下にあるAlgorithm parametersを開いて、PSI/PHI/DELTA BLASTの「PSI-BLAST Threshold」を0.0001に変更します。この値は小さくするほど偽陽性が少なくなり、学術論文では0.0001の値が使われることが多いです。
この条件でBLASTを実行した結果がこちらです。
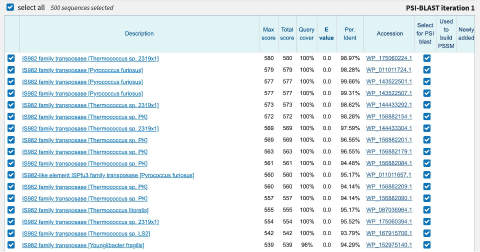
このように、データベースに登録されている多くのホモログを取ることができます。アノテーションを見てみるといずれもtransposaseとついていることが分かります。
これらのたくさんの配列をもとにアラインメントし、Profileを作成してBLASTを行うには、下側にあるこのボタンをクリックします。

すると、1回目のBLASTでは検出できなかったホモログが追加で取得できます (黄色)。
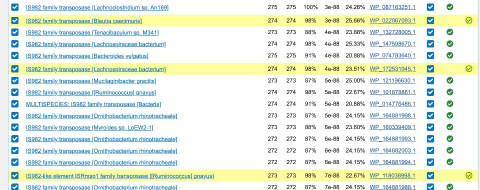
これを4 ~ 5回繰り返していくことで、遠いホモログ遺伝子を拾い上げていく方法がPSI-BLASTです。
関連図書
この記事に関連した内容を紹介している本やサイトはこちらです。
Biopython入門 - 中編 【NCBIデータベースやBlastへのアクセス】
今日も【生命医学をハックする】 (@biomedicalhacks) をお読みいただきありがとうございました。当サイトの記事をもとに加筆した月2回のニュースレターも好評配信中ですので、よろしければこちらも合わせてどうぞ