RNAは多数のRNA結合タンパク質 (RBPs) と共同して生命機能を担っています。
この記事では、その相互作用を研究する方法であるCLIP-seqと関連する方法について、参考文献と一緒に見ていきます。
この記事の内容
CLIP-seqができるまで
次世代シークエンサーと質量分析によるプロテオミクスの技術的進歩により、これらのRNAとRBPの相互作用の全体像を分析できるようになりました。
初期には、ランダム配列RNAのライブラリーを使ってRBPが結合する配列を調べることができるSELEXが開発されました(In vitro selection of RNA molecules that bind specific ligands. Nature 1990)。
RNAとタンパク質の相互作用をまとめて解析するために広く受け入れられた最初の方法は、HITS-CLIP (CLIP-seqとも) でした。
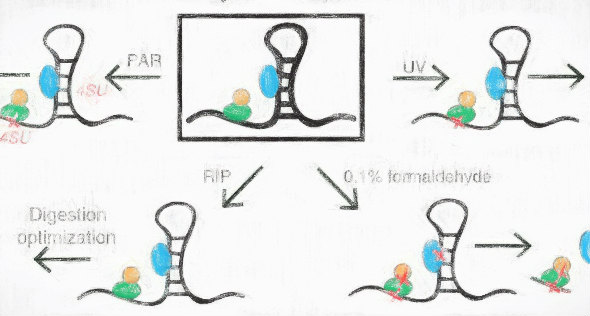
254 nmの紫外線 (UV) を照射することでタンパクとRNAを架橋した後に、目的のタンパクに対する抗体で免疫沈降 (crosslinking and immunoprecipitation, CLIP)し、当時は新しかったRNA-seqによって知りたいタンパクが結合しているRNAを網羅的に探索する方法です(HITS-CLIP yields genome-wide insights into brain alternative RNA processing. Nature 2008)。
この技術の中心はUVによる架橋であり、これは生きた細胞で行うことができて、しかもタンパク-タンパク質よりもはるかに高い効率でRNA-タンパク質相互作用を架橋するできるというものでした。
目的のタンパクが結合するRNAを調べる方法
クロスリンクする方法
最初にHITS-CLIP法が発表されてから、クロスリンクを365 nmで行うPAR-CLIP法 (Transcriptome-wide identification of RNA-binding protein and microRNA target sites by PAR-CLIP. Cell 2010)、cDNAのライブラリー調製を工夫したiCLIP法(iCLIP reveals the function of hnRNP particles in splicing at individual nucleotide resolution. Nat. Struct. Mol. Biol. 2010)、免疫沈降に細工があるCRAC法(Identification of protein binding sites on U3 snoRNA and pre-rRNA by UV cross-linking and high-throughput analysis of cDNAs. PNAS 2009)といった改変版の方法が次々に登場しました。
これらはいずれもRNAとタンパクをクロスリンクしてから解析するという点で共通しています。
クロスリンクしない方法
クロスリンクをせずにRNA結合タンパク (RBP) を探索する方法も考案されています。
TRIBEという方法は、目的のRBPにアデノシンデアミナーゼ (ADAR) をつけて細胞に導入します。
ADARがRNAのアデノシンからイノシン に編集する性質とその後の逆転写中にイノシンが主にCに変換されることを利用して、RNAを回収し逆転写後の産物を大規模シークエンサーでマッピングすることでどこに結合していたのかをコンピューター計算で求めます (TRIBE: hijacking an RNA-editing enzyme to identify cell-specific targets of RNA-binding proteins. Cell 2016)。
より編集活性の強いADAR変異体を使ったHyperTRIBEも発表されました (Mechanistic implications of enhanced editing by a HyperTRIBE RNA-binding protein. RNA 2018)。
RNA taggingという別の方法もあります。線虫のポリ (U) ポリメラーゼであるPUP‐2を調べたいRBPへ融合させたものを細胞に導入します。
RBPとRNAが近接すると、PUP-2がRNAをポリU化する結果、ポリUを濃縮してシークエンサーにかければどのRNAと相互作用がわかるという手法です (Protein-RNA networks revealed through covalent RNA marks. Nat. Methods 2015)
。
いずれもIPなどでRNA-タンパク複合体をとってくる必要はないというメリットがありますが、融合タンパクのコンストラクトを作らないといけないというデメリットもあります。
関心のあるRNAに結合するタンパクを同定する
これまでは、タンパクに結合するRNAを知りたいという問題に対する研究手法を紹介しましたが、当然逆もあります。
つまり、「RNAに結合するタンパクを知りたい」という場合です。
知りたいのはタンパクなので、質量分析計を使うことになりますが、次世代シークエンサーと比べロースループットであり、またタンパクは核酸のようにたやすく増幅できないのでこちらの方が難易度は高くなります。
最もストレートな方法の1つとして、RNAとタンパクを紫外線を当てることで架橋させ、オリゴdTビーズを使ってポリA付加されている複合体を集めたあと、酵素でRNAを消化してから質量分析計にかけるという方法が取られてきました (Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell, 2012)。
しかしこの方法ではポリAが付加されるRNAしか濃縮できません。
ポリA付加されていないRNAでも結合タンパクを調べることができるように、まずRNAを5-ethynyluridine (5-EU) でラベルしておいて、5-EUにクリックケミストリーでビオチンを結合させ、それをストレプトアビジンビーズで集めてからRNA消化 & 質量分析計という流れをとるRICK(Capturing the interactome of newly transcribed RNA. Nat. Methods 2018)やCARIC (Transcriptome-wide discovery of coding and noncoding RNA-binding proteins. PNAS 2018) と呼ばれる方法が開発されました。
酸性のグアニジニウムチオシアネート/フェノール/クロロホルム (トリゾールなど) を使ったRNA抽出 (RNAは水層だがDNAとタンパクは有機層にくる) はよく使われている手法ですが、RNA-タンパクを架橋したものは通常は捨てている中間層にいることが分かりました。
この中間層を回収してRNA-タンパク複合体を解析する手法が2019年に3つ報告されています。
XRNAX (The human RNA-binding proteome and its dynamics during translational arrest. Cell 2019)
OOPS (Comprehensive identification of RNA-protein interactions in any organism using orthogonal organic phase separation (OOPS). Nat. Biotechnol. 2019)
pTEX: (Purification of cross-linked RNA-protein complexes by phenol-toluol extraction. Nat. Commun. 2019)
関連サイト・図書
この記事に関連した内容を紹介しているサイトや本はこちらです。
まとめ
最後に今回の内容をまとめます。
- RNAとタンパクの相互作用を検出ベースはCLIP-seq
- クロスリンクしない方法も考案されている
- 質量分析計を使ってRNAと結合するタンパクを同定することもできる
今日も【医学・生命科学・合成生物学のポータルサイト】生命医学をハックするをお読みいただきありがとうございました。