NCBIは生命科学系のさまざまなデータを集積していますが、その公式のゲノムビューワーがGenome Data Viewerです。この記事では、Genome Data Viewerの使い方をいくつかの例とともに紹介します。
この記事の内容
NCBI Genome Data Viewerの基礎的な使い方
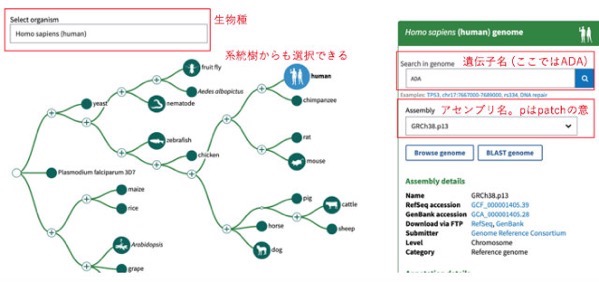
Genome Data ViewerはNCBIのメインゲノムブラウザーになりました (以前はMap Viewerだった)。まずはホームページにアクセスします。
遺伝子名を入力 (ここではADA, Adnosine Deaminase)し、Assemblyを選択 (ここではGRh38.p13) を選択し検索ボタンをクリックします。
そうするとこのような画面になります。上段には染色体の絵やGene & Exon Navigator、左側には各種検索ツールがあります。
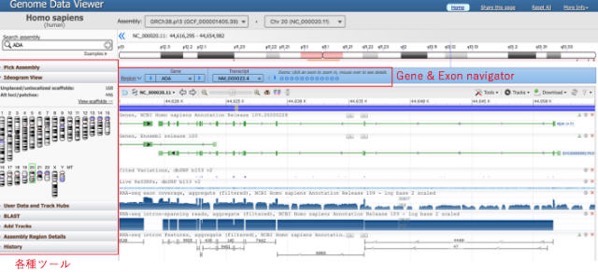
ADA遺伝子にmutationが入ると重篤な免疫不全症になることが分かっています。そこでそれらの臨床情報をここに表示してみます。
メインパネルの右上にある「Tracks」> 「NCBI Recommended Track Sets」 > 「Clinical」を選択します。
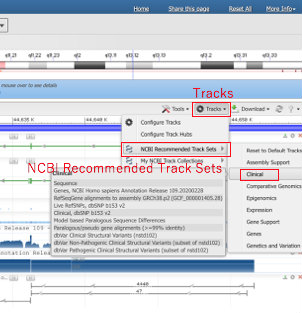
そうするとdbSNPや臨床的に意義のあるSNP、あるいはもっと大きな構造異常などが表示されてきます。もちろん不要なトラックは右上の赤い☓をクリックすることで消すことが可能です。

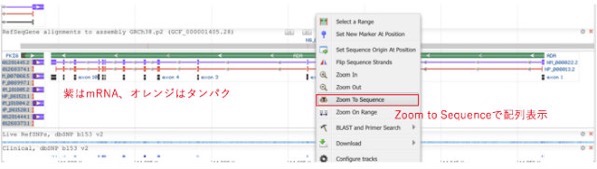
メインディスプレイに表示されているもののうち、紫は転写産物でオレンジがタンパクです。エクソンを選択して右クリック「Zoom to Sequence」でその場所までズームすることができます。
Minor Allele Frequency (MAF) > 0.01のSNPを表示してみます。「Track」から「Configure Tracks」を選択し、dbSNPにチェックを入れます。
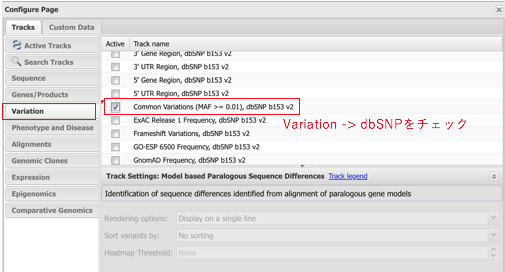
そうするとこのようにどの塩基にどのようなSNPがあるのかを調べることができます。
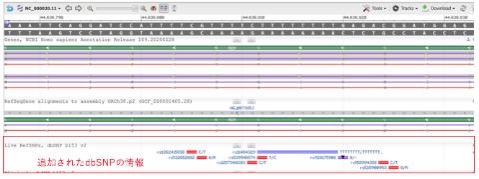
自分でデータを持っていれば左側のメニューにある「User Data and Track Hubs」からアップロードしゲノムブラウザに表示することもできます。
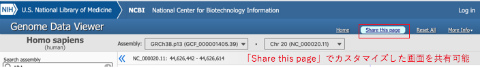
ゲノムブラウザの右上にある「Share this page」を押して出てくるURLを使えばこのページを他の人とシェアできます。
NCBI Genome Data Viewerを経由してプライマーをつくる
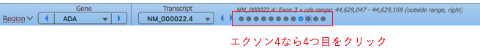
上部にあるExon navigatorを使えば個々のエクソンを簡単に移動できます。例えばExon 4に移動したいときには、(この遺伝子は逆の鎖にのっているので) 右から4つ目をクリックします。
それではこの領域をPCRで増幅するためのプライマーを作ってみます。「Tools」 > 「Primer BLAST」(Visible Range)を選択します。
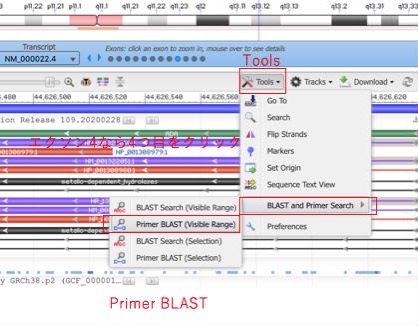
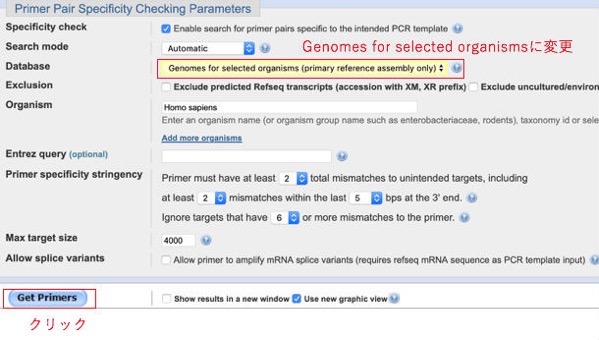
するとNCBIのツールの1つPrimer-BLASTのページになり、必要事項が記入された状態になっています。初期状態ではmRNA配列を増幅するようになっているので、DNAを増幅するために「Database」から"Genomes for selected organisms"を選択し「Get Primers」ボタンを押します。
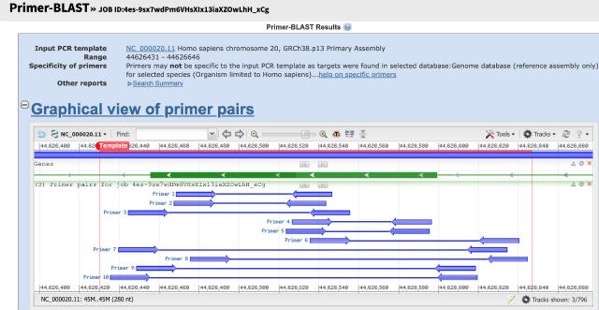
1分程度してから、このような結果のページが表示されます。
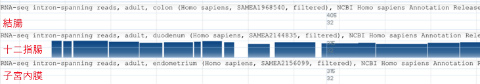
Genome Data Viewerから組織ごと・エクソンごとの遺伝子発現量を調べる
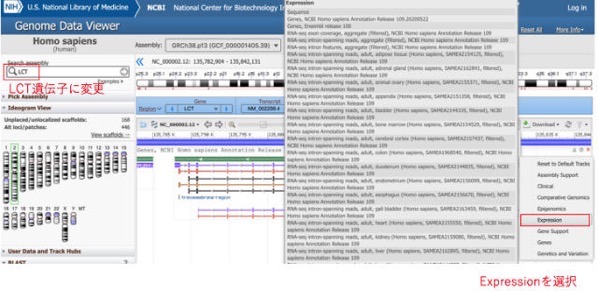
今度はLactase (LCT) を検索してみます。もし遺伝子発現量に興味がある場合には、「Tracks」「NCBI Recommended Track Sets」から「Expression」を表示させることができます。
上段にはさまざまな臓器の発現を全部まとめた図が示されてますが、

個別臓器については下段に表示されていて、lactataseが十二指腸で強く出ていることが分かります。
関連図書
この記事に関連した内容を紹介している本はこちらです。
今日も【生命医学をハックする】 (@biomedicalhacks) をお読みいただきありがとうございました。
当サイトの記事をもとに加筆した月2回のニュースレターも好評配信中ですので、よろしければこちらも合わせてどうぞ