RNA-seq解析は今や多くの生命科学研究に取り入れられており、その基本的なデータ解析については自分で一通りできた方がよい状況になっています。
このRNA-seqデータ解析を手助けしてくれるwebツールの1つがiDEPであり、さまざまなことをこのツールのみで完結することができます。
データのアップロードと前処理
まずiDEPのサイトにアクセスします。
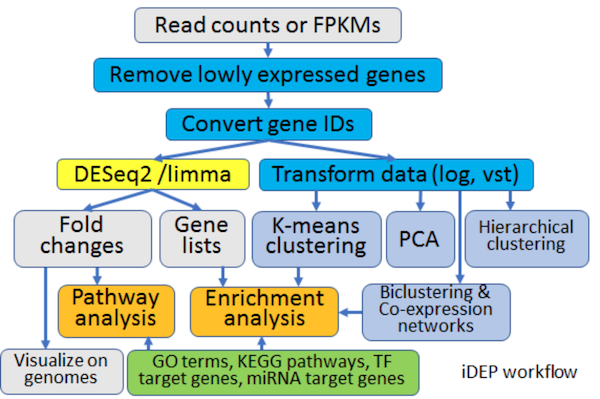
iDEPはRNA-seqにおける一通りのデータ解析ができるウェブツールで、このような流れで進めていきます。
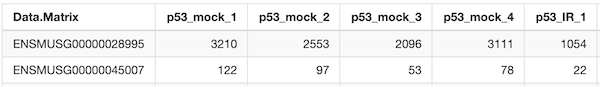
iDEPには、原則としてカウントデータをアップロードします。具体的には、データの1列目にはgene ID, 1行目はサンプル名を記載した2x2の形のカウントデータを用意します。
具体的にはこのような形になります。
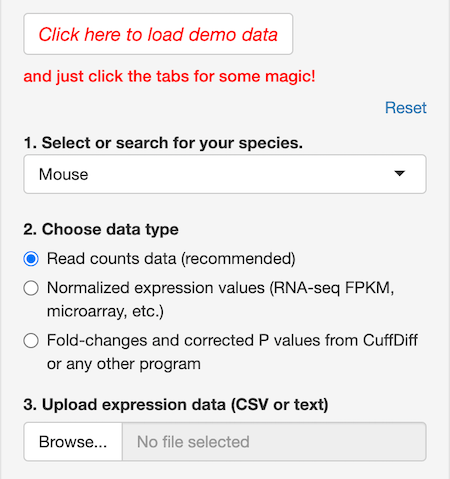
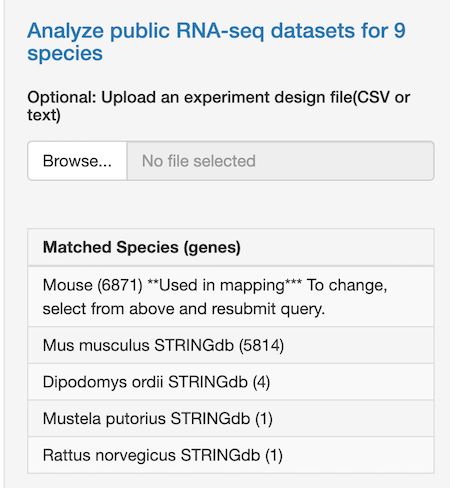
これをアップロードし、生物種なども指定します。
その下側にある「Analyze public RNA-seq datasets」というところにアクセッション番号を入れれば、既存のRNA-seqデータを再解析することも容易にできます。
具体的にはARCHSやDigital Expression Explorer 2 (DEE2)に登録されている、NCBI等にアップされた次世代seqデータを統一のパイプラインで事前処理しているデータを使っています。
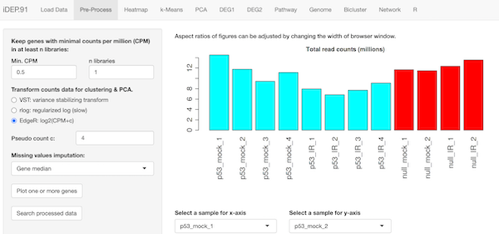
データが正常に読み込まれたら、次にPre-processタブに移ります。ここでは低発現遺伝子のフィルタリングやリードカウントの正規化を行います。
CPMは100万リードあたりのカウント数のことで、ここではそれぞれのサンプルで100万リードあたり0.5カウントもないような低発現遺伝子については除くという設定をしています。この値はもちろん自由に変更することができます。
遺伝子のクラスタリング
発現変動遺伝子群の視覚化は、次のHeatmapタブで行えます。ヒートマップは全ての発現変動遺伝子を使って描かれているのではなく、発現変動を表す標準偏差SDが大きい上位 (デフォルトでは1000) 遺伝子のみを使っています。
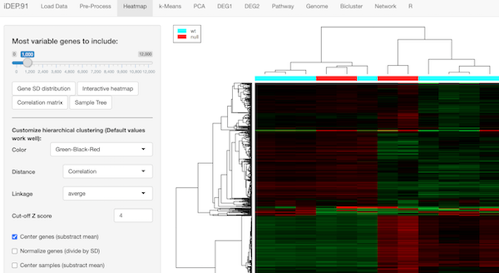
その下側にある「Gene SD distribution」をクリックするとSDの分布がどのようになっているのかを確認できるので、これを使ってどれくらいの遺伝子をヒートマップに使うのか考えることができます。
このヒートマップで使われている階層的クラスタリングは感度がやや低く、グループにはっきり分けられないことも少なくありません。しかも扱う数が増えると計算負荷が大きくなります。
よく使われる他のクラスタリング手法として、K-means法も利用可能です。デフォルトではSDが大きいトップ2000の発現変動遺伝子を4つのクラスタに分けることになります。
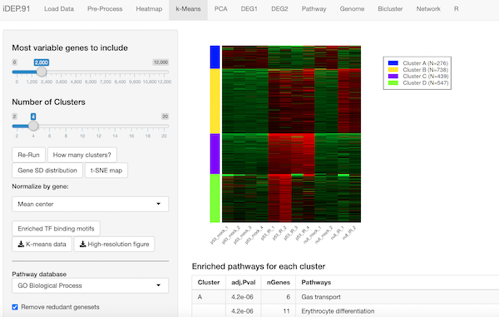
さらに、それぞれのクラスターにおけるGO等のenrichment解析や、どのような転写因子結合モチーフが濃縮しているかを調べることもできます。
発現変動遺伝子の抽出
DEGのタブでは、統計的に有意なDEGを絞り込めます。Fold ChangeやFDRのカットオフ、解析に使うツール (DESeq2など) を選択できます。
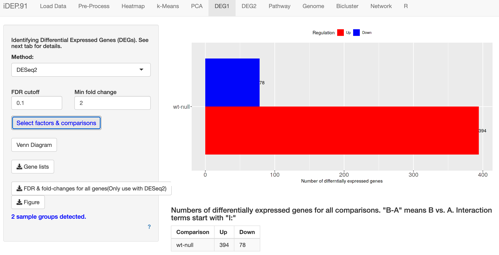
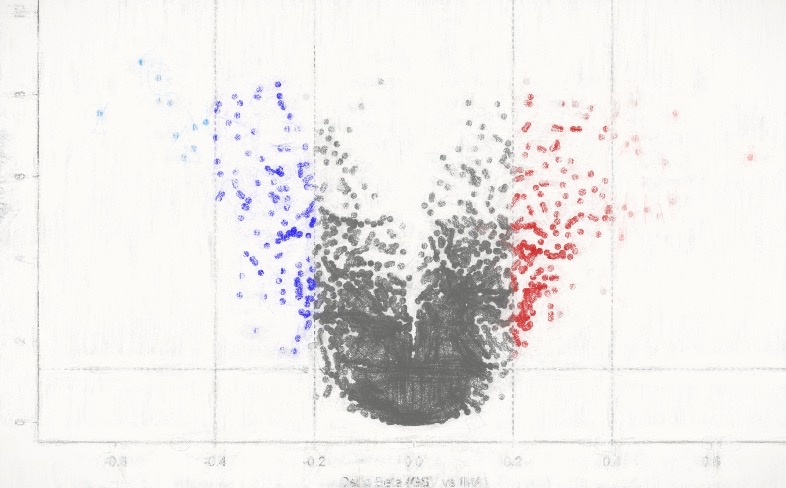
Volcano plotなど、さまざまなプロットを表示したり、発現上昇する遺伝子、発現低下する遺伝子についてどのようなものがあるのかGO解析や転写因子結合モチーフ解析などが可能です。
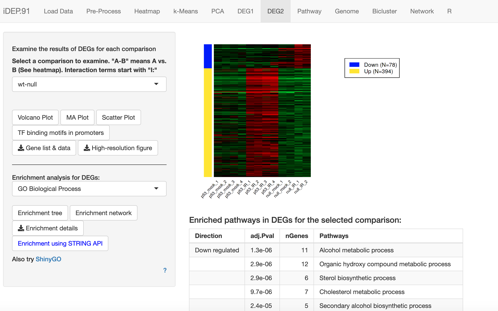
その他の解析
ここまではRNA-seqの定番解析を見てきましたが、他にもさまざまなことがiDEPでできます。
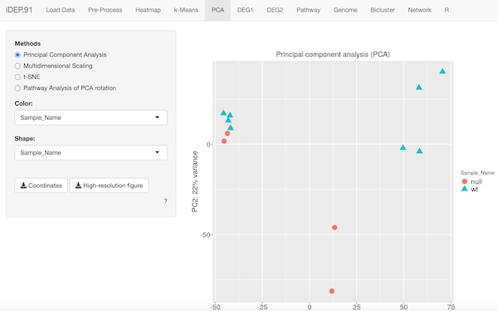
例えば、主成分分析 (PCA)、多次元尺度構成法 (MDS), t-SNEなども可能です。
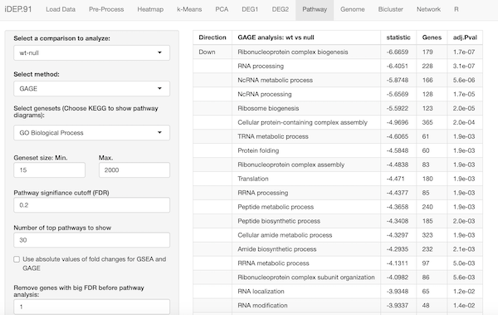
発現が落ちている (あるいは上がっている) 遺伝子はどのようなパスウェイのものが多いのかや、ネットワーク解析もできます。
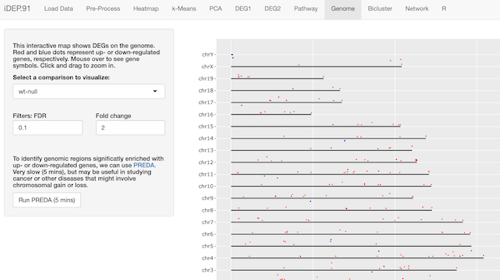
ちょっと珍しいものでは、ゲノム内のどの部位に発現変動遺伝子が多いのかを調べることもできます。
また、iDEPで得られた計算結果を再現するためのRコードも自由にダウンロードすることができるようになっています。iDEPでいろいろパラメーターを動かしながら直感的に操作し、そのRコードをダウンロードして、さらに細かい解析をすることができます。
RNA-seqにおけるRの利用については、Useful Rシリーズなどに紹介されています。
まとめに代えて
この記事では、RNA-seqの代表的なテータ解析を行うためのwebツールiDEPを紹介しました。
似たようなツールは他にもいろいろありますが、iDEPはこれだけでほとんど全てのことができること、そして使いやすく見やすい設計になっていることもあってかなり使用されるツールになっています。
TogoTVには分かりやすい動画説明もあり、
「RNA-Seqデータ解析 WETラボの鉄板レシピ」という本には詳しい使い方の説明も書かれています。
もはやRNA-seqデータ解析の基本は生命科学系の研究をする上で避けては通れません。iDEPにぜひ触れてみてください。
今日も【生命医学をハックする】 (@biomedicalhacks) をお読みいただきありがとうございました。当サイトの記事をもとに加筆した月2回のニュースレターも好評配信中ですので、よろしければこちらも合わせてどうぞ









