過去10年の間に、Golden Gate法やGibson assembly法などの重要な技術が開発されました。
この記事では、やや複雑なプラスミドを構築するときに知っておきたいそれらアセンブリ法をまとめました。
プラスミドってそもそも何という方はプラスミドの基礎 【よく使うプラスミドベクターも】にまとめていますのでそちらをお読みください。
この記事の内容
制限酵素ベースのアセンブリー法
BioBrick
BioBrickは、クローニング用に最初に開発されたプラットフォームの1つで (Engineering BioBrick vectors from BioBrick parts. J. Biol. Eng. 2008)、4種類の制限酵素のみを使用して何度でも,新たなDNAを付加することが可能になっています。
すでにさまざまなパーツが4種類の制限酵素サイト (EcoRI, SpeI, Xba I, Pst I)を両脇に持つように組み込まれていて、これらの制限酵素はどれで切っても切り口が同じになるので、ライゲーションで繋げられるというのが基本的な仕組みです。
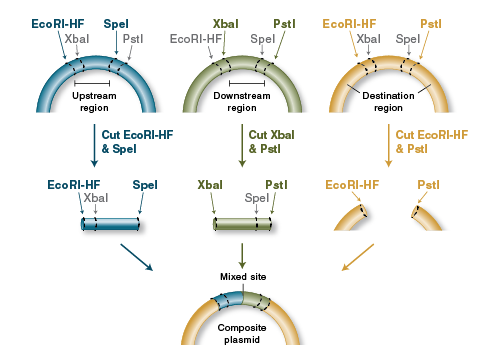
https://international.neb.com/applications/cloning-and-synthetic-biology/dna-assembly-and-cloning/biobrick-assembly
BioBrickのパーツはたくさんリポジトリーに登録されていて、これらを自由に利用できるだけでなく、iGEMという合成生物学の学生世界大会でも採用されています。
BglBrick
BioBrickプラットフォームが広く採用されるようになると、これをベースとしてより効率が向上し便利になった、さまざまな新しいプラットフォームも開発されるようになりました (BglBricks: a flexible standard for biological part assembly. J. Biol. Eng. 2010)。
BglBrickはその代表例で、BglとBamHIの制限酵素で切り出すと、それらのoverhangを繋げることができ、しかもこの場合にできるグリシン-セリンはほとんどの場合に無害なリンカーとして働くので融合タンパクも簡単に作れるという系です。
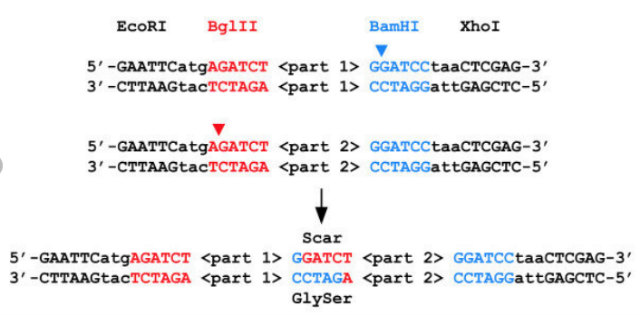
https://www.researchgate.net/figure/Standard-assembly-of-BglBrick-parts-Unique-BglII-in-red-and-BamHI-in-blue_fig1_41806403
breadboardやSEVA
複数のプラスミドコンストラクト中のパーツの交換を容易にするために開発された方法もあります。
その代表例の1つがbreadboardプラットフォームで、これは大腸菌遺伝子ネットワークコンストラクトの迅速な設計のために作られました (Iterative plug-and-play methodology for constructing and modifying synthetic gene networks. Nat. Methods 2012)。
細菌用のプラスミド構築支援をしてくれたり、プラスミドごとのデータを集めた網羅的なリソースでもあるオンラインツールStandard European Vector Architecture (SEVA)は2009年に発表され今では合成生物学界隈で広く使われており、統一のルールにしたがったプラスミド情報共有に大きな役割を果たしています。
https://youtu.be/I8gZjXDRQ2I
iBrick, HVAS
制限酵素による消化とライゲーションという古典的な手法の欠点は、クローニングする前に配列内の不必要な制限酵素認識部位を潰しておかないといけないことです。
I-SceIのようなホーミングエンドヌクレアーゼは、認識配列が長いためほとんど認識部位がないので、この問題を克服する方法の1つとして提案されています (Homing endonucleases: from basics to therapeutic applications. Cell. Mol. Life Sci. 2010)。
実際、BioBrickプラットフォームによく似ているiBrickプラットフォームでは、ホーミングエンドヌクレアーゼ認識部位を含むプレフィックスおよびサフィックス配列を使います (iBrick: a new standard for iterative assembly of biological parts with homing endonucleases. PLoS ONE 2014)。
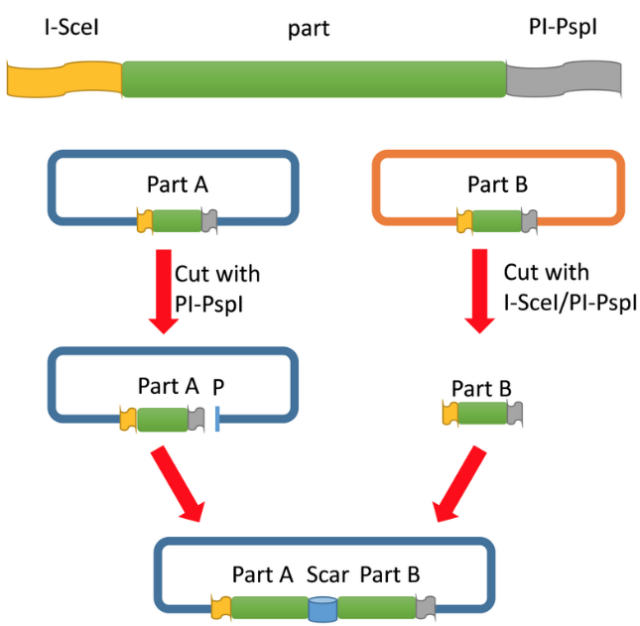
https://www.researchgate.net/figure/Schematic-diagram-of-iBrick-forward-assembly-A-Construction-of-iBrick-parts-which_fig7_267102259
HomeRun Vector Assembly System (HVAS)もホーミングエンドヌクレアーゼを利用していますが、こちらは多数のインサートを同時に入れることが可能なフレームワークです。
Golden Gate
IIS型制限酵素は、通常のII型制限酵素とは異なり、認識部位から数塩基離れたDNAを切断します。これを使うと、「シームレス」なDNAの組立てができるという大きな利点があります。しかも1つの酵素で異なる突出部をもつ断片を生成できるので、これらをたくさん一度に決められた順序で連結することもできます。
これを応用したのがGolden Gate法です。すでにNEB社などから商品化されており、原理についての分かりやすい動画もあるのでここに掲載しておきます。
動画の中ではIIs型制限酵素の例としてBsaIが使われていましたが、他にもたくさんのIIs型制限酵素があります。
Golden Gateに使うパーツは、PCRや遺伝子合成で用意できます (Precise and reliable gene expression via standard transcription and translation initiation elements. Nat. Methods 2013) が、アセンブリ時のエラーを最小にするために一度エントリーベクターに入れ、それを切り出してつなぎdestination ベクターを作るという方法がとられています。
MoClo, mMoCloプラットフォーム
もともと植物分子生物学用に開発されたMoClo (GoldenGate Modular Cloning)プラットフォームを使えば、Golden Gateを1度に階層的に実施することができますが、それには多数のエントリーベクターとデスティネーションベクターが必要になるという弱点もあります(A modular cloning system for standardized assembly of multigene constructs. PLoS ONE 2011)。
https://youtu.be/_bag7cdwZpY
GoldenBraidプラットフォームでは、遺伝子アセンブリをペアで行うことで必要なベクターの数を減らすことができますが、その反面として大きなコンストラクトを作るときにはより多くの工程が必要になります(GoldenBraid: an iterative cloning system for standardized assembly of reusable genetic modules. PLoS ONE 2011)。
これらを解決したのがmammalian MoClo (mMoClo)プラットフォームです。このプラットフォームでは、非常に大きな、かつたくさんの遺伝子からなるコンストラクトを哺乳類の染色体の特定の部位に安定に組み込むことができます (A platform for rapid prototyping of synthetic gene networks in mammalian cells. Nucleic Acids Res. 2014)。
GreenGate, MASTER
古典的な制限酵素によるクローニングの場合と同じく、Golden Gateは制限酵素認識配列がDNA配列に他に存在しないということが必要です。
この制限を克服するために、制限酵素で切断されなくするためにDNAメチル化を応用する仕組み (GreenGateやmethylation-assisted tailorable ends rational (MASTER) ライゲーション法など) も使われ始めました(GreenGate — a novel, versatile, and efficient cloning system for plant transgenesis. PLoS ONE 2013 ; The MASTER (methylation-assisted tailorable ends rational) ligation method for seamless DNA assembly. Nucleic Acids Res. 2013)。
組み換えによるアセンブリ
Gateway
ファージのインテグラーゼとattachment (att) 配列を使った部位特異的な組み換え法も考案されています。その代表がGatewayシステムです。
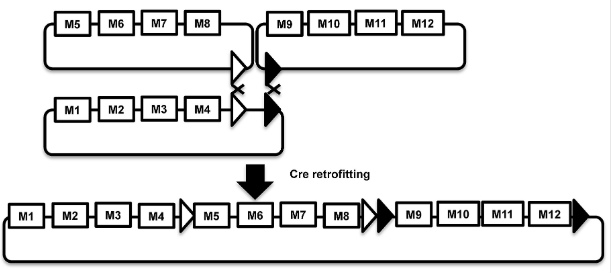
https://figshare.com/articles/_HomeRun_Vector_Assembly_System_A_Flexible_and_Standardized_Cloning_System_for_Assembly_of_Multi_Modular_DNA_Constructs_/1082260/1
λインテグラーゼを使ったGatewayシステムでは、興味がある遺伝子の両脇にattBサイト (attB1とattB2) を用意したentry vectorと、移し替えたい目的のdestination vector上のattPサイト (attP1とattP2) の間の組み換えを利用して、正確にクローニングすることができます (DNA cloning using in vitro site-specific recombination. Genome Res. 2000)。
YouTubeに分かりやすい解説があるので紹介します。
https://youtu.be/qGSI5qxPDM0?t=66
最近では、4つのatt組換え配列を合成することで、複数のパーツを同時にクローニングできるように拡張されたGatewayシステムもあります (Evidence for high specificity and efficiency of multiple recombination signals in mixed DNA cloning by the multisite Gateway system. J. Biotechnol. 2004)。
反応はとても簡単で、試薬を混ぜて1時間待つだけです。
https://youtu.be/wHcaKnAkEag
RMCE
RMCE (Recombinase-mediated cassette exchange) とは、Cre-loxP系などのシステムを利用して、プラスミド間でカセットを交換する一連の方法です。
複数のプラスミド間を、このように連結することもできます。
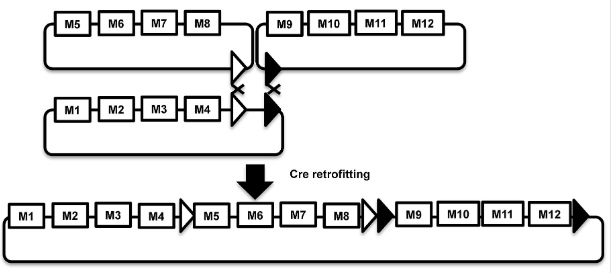
長いオーバーラップを活用したアセンブリ法
Gibson
長い塩基配列の相同性 (20~40 bpがよく使われる)があると、DNAアセンブリの効率や特異性が向上します。Gibsonアセンブリがその代表だということは別の記事で紹介しています。
fa-arrow-circle-right関連記事クローニングの方法まとめ 【制限酵素だけではない】
この方法を使えば、1ステップで5個以上のパーツを容易に組み上げることができ、これはより大きなDNA断片(1kb以上)を操作するのに特によく用いられています。
生物の組み換え活性を利用する方法
枯草菌 (Bacillus subtilis) や 出芽酵母 (Saccharomyces cerevisiae) のような生物が持つDNA修復機構を応用して、つまり天然に起こる相同組換えによって、in vivoで目的のコンストラクトを組み立てる方法もあります (Bottom-up genome assembly using the Bacillus subtilis genome vector. Nat. Methods 2008 ; One-step assembly in yeast of 25 overlapping DNA fragments to form a complete synthetic Mycoplasma genitalium genome. PNAS. 2008)。
これらの生物は相同組み換え活性が高いことが知られていて、相同な領域を持つDNAを入れると中で繋がるのです。
DNAアセンブリーをもっと身近に
一昔前は、DNAコンストラクトを作るのがとても大変だったそうですが、技術の進歩のおかげで、今や大学の学部生でさえ、真核生物の染色体全体を作りあげることもできます(Total synthesis of a functional designer eukaryotic chromosome. Science 2014)。
特にアメリカでは研究者でない「アマチュア科学者」によるDIYバイオの機運も高まっています。
fa-arrow-circle-right関連記事DIYバイオの夜明け【インスリンを自宅で作る】
この記事で紹介した技術の中に1つでも初めて知ったものがあればぜひ試してみてください。
関連サイト・図書
この記事に関連した内容を紹介しているサイトや本はこちらです。
CRISPR-Casによるゲノム編集 【2010年代の総まとめ】
まとめ
最後に今回の内容をまとめます。
- Golden Gate, Gibson, Gatewayだけは覚えておく
- さまざまな技術が日進月歩で開発されている
- 大学の学部生でも染色体を作成できる
今日も【医学・生命科学・合成生物学のポータルサイト】生命医学をハックするをお読みいただきありがとうございました。