目的の遺伝子を増やすことをクローニングといい、生命科学研究の基本の1つです。一昔前は、制限酵素といういわば「はさみ」を使って、切り口の対応関係を利用してクローニングすることが普通でした。
近年では、制限酵素以外のクローニング手法もたくさんあります。この記事では、生命科学系の学生さん向けに、そのような「発展的な」クローニング手法の概略とその文献、有用なリソースを紹介します。
この記事の内容
クローニング法の誕生
1970年初頭、制限酵素HindIII (A restriction enzyme from Hemophilus influenzae. I. Purification and general properties. J. Mol. Biol. 1970)やEcoRI (R factor-controlled restriction and modification of deoxyribonucleic acid: restriction mutants. J. Bacteriol. 1972) を皮切りに次々と発見され、組換えDNA技術の開発に大きな突破口が開かれました。
プラスミドを使って、ある細菌株から別の細菌株にDNA断片を移す最初のクローニング実験が行われたのは1973年のことです (Construction of biologically functional bacterial plasmids in vitro. PNAS 1973)。
異なる種由来のDNAをクローン化し、別の種で増やすことができるという発見 (Replication and transcription of eukaryotic DNA in Escherichia coli. PNAS 1974)は、遺伝子組換え技術の始まりを意味しました。
1980年代には、PCRによりDNA断片をin vitroで増やせるという画期的な発明 (Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science 1988) によって、高度なクローニング法がたくさん出現しました。
制限酵素とライゲーションによるクローニング手法
現在、制限酵素は200種類以上のものが高度に精製された状態で市販されています。
多くの制限酵素は線状PCR産物をうまく切断できず (環状DNAを好むことが多い)、また「シームレスな」 クローニングは必ずしも可能ではないという弱点もあります。
そこでいろいろな方法が考案されました。例えば、認識配列と切断配列が違うIIs型制限酵素は、シームレスなクローニングを行うことができます (Creating seamless junctions independent of restriction sites in PCR cloning. Gene 1996)。
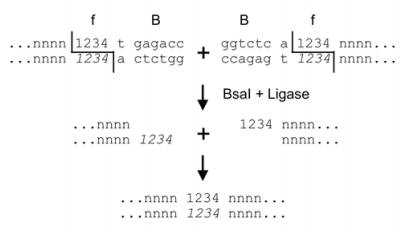
Source: https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0005553
このアプローチはGolden Gate cloning (Golden gate shuffling: a one-pot DNA shuffling method based on type IIs restriction enzymes. PLoS ONE 2009) として採用され、現在ではNEBによって商品化もされています。
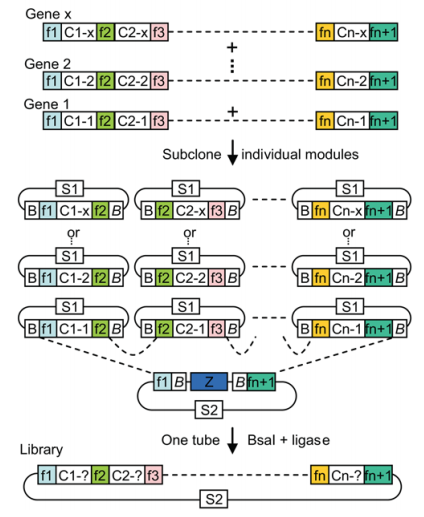
Source: https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0005553
ThermoFisherが商品化しているTOPO-TA(sub) cloningは制限酵素を使わないクローニング法です。
これはTaqポリメラーゼの特異的な性質を利用しています。Taqポリメラーゼは二本鎖DNA断片に3'突出したアデニン (A) 塩基を付加するので、5’末端に突出したチミジン (T) を持つようなベクターを用意しておけばライゲーションが効率的になります。
別のUSER cloningという方法は、特別な酵素が必要ではあるものの一方向性でクローニングできます。まずDNA断片を、5’末端に特異的7 bp配列を持ちデオキシウラシル(dU)塩基があるプライマーで増幅します。
この配列は、USER酵素によって認識・切断され、8 bpの5’突出部を有する断片を残すので、方向性を持ってベクターにクローニングすることができるという方法です (USER friendly DNA engineering and cloning method by uracil excision. Nucleic Acids Res. 2007)。
NEBからキットが出ています。
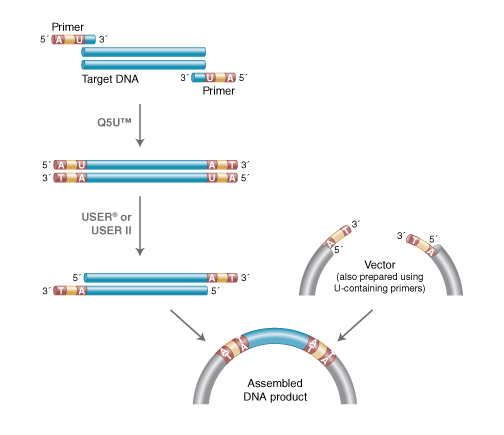
Source: https://international.neb.com/applications/cloning-and-synthetic-biology/user-cloning
Ligation-Independent Cloning (LIC) では、PCR断片と線状化ベクターはいずれも~12~18bpの相補的配列に接していて、四つのヌクレオチド(dATP、dCTP、dGTP、dTTP)のうちの一つを欠いた状態で反応を行います (Ligation-independent cloning of PCR products (LIC-PCR). Nucleic Acids Res. 1990)。
一本鎖DNAオーバーハングは、そこに存在しない特定のヌクレオチドのみがあるという条件で、PCR断片およびベクターをT4ポリメラーゼと別々にインキュベートすることによって作製できます。通常は校正機能を果たすポリメラーゼが持つエキソヌクレアーゼ活性は、反応に加えられた特定のヌクレオチドが必要になるまで3'部位からヌクレオチドを除去していくので、効果的に長い一本鎖突出部を形成することが可能です。
PCR断片とベクターを混合し、相補的な付着末端をアニーリングさせると、オーバーハングが大きいために大腸菌細胞内でligationすることが可能です。
このLICクローニングは簡単に実施でき、比較的安価な試薬のみでいいので、一般的な技術となりました。
特定の相補的配列が必要だという制限は、Sequence and Ligation-Independent Cloning (SLIC)では克服されています (Harnessing homologous recombination in vitro to generate recombinant DNA via SLIC. Nat. Methods 2007)。
T4ポリメラーゼによる処理はヌクレオチドがない条件下で行われるので、より広範な一本鎖突出ができるからです。
Gibsonらは、LICとSLICの利点を組み合わせて、配列に制限なく、しかも短い (15bp) 相補的DNAでよいクローニング手法を開発しました (Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat. Methods 2009)。T5エキソヌクレアーゼで処理した後にポリメラーゼとリガーゼを添加するという手法です。
Gibsonクローニングのための試薬は、NEBから市販されています (Gibson Assembly cloning kit)。
他の企業も同様のクローニングキットを提供していて、In-Fusion Cloning (タカラバイオ)、GeneArt Seamless Cloning(サーモフィッシャー)、Cold Fusion Cloning (SBI)などがあります。
組み換えベースのクローニング
Cre-loxPや相同組み換えなど、組み換えベースの方法もあります。これらの方法はすべて、Entry ベクターとDestinationベクターという2つのベクターが必要です。
Univector plasmid–fusion system (UPS) は2つの異なるベクターにあるloxPサイトがCreにより融合することを利用してコンストラクトを作る方法です(The univector plasmid-fusion system, a method for rapid construction of recombinant DNA without restriction enzymes. Curr. Biol. 1998)。
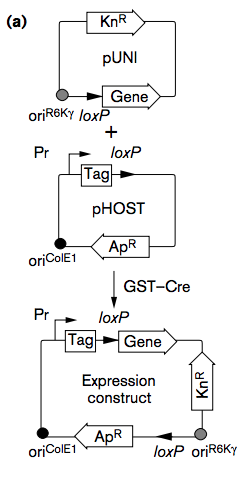
Source: https://www.sciencedirect.com/science/article/pii/S096098220700560X?via%3Dihub
Gatewayシステムは、ラムダバクテリオファージがインテグラーゼ (Integrase) とatt 組換え部位によって大腸菌ゲノムへ組み込まれることを利用したシステムで、これを使えばattBサイトを含むPCR断片をドナーベクターやdestinationベクターに直接移し替えることが簡単にできます(Single step BP/LR combined Gateway reactions. Biotechniques 2013)。
SLiCE(Seamless Ligation Cloning Extract)クローニングという手法は、相同な配列を持つベクターとインサートのラーゲーションを大腸菌細胞抽出物を使って行うというユニークな方法です (SLiCE: a novel bacterial cell extract-based DNA cloning method. Nucleic Acids Res. 2012)。
相補的な部分の長さについても検討されていて、その結果、少なくとも15 bp の相補的な領域が必要であり、長いほうがいいという結論が得られています。
PCRベースのクローニング
PCRによるクローニング法は、ベクターやインサートを一緒にPCRで増幅する必要がありますが、その代わり制限酵素やligaseなどを必要としません。
Polymerase Incomplete Primer Extension (PIPE) クローニング (Combining the polymerase incomplete primer extension method for cloning and mutagenesis with microscreening to accelerate structural genomics efforts. Proteins 2008) は、PCR反応中にそれぞれの断片が不完全な伸長され、異なる長さの一本鎖突出部を持つ混合物ができるという事実に基づいています。14~17bpの相補的なオーバーハングを持つインサートとベクターは、両方ともそれぞれPCR反応で増幅し、PCR産物を混合して大腸菌などに形質転換するとつながったコンストラクトが取れるという手法です。
Restriction-Free (RF) cloning (RF cloning: a restriction-free method for inserting target genes into plasmids. J. Biochem. Biophys. Methods 2006) やOverlap Extension PCR cloning (Overlap extension PCR cloning: a simple and reliable way to create recombinant plasmids. Biotechniques 2010) は、Quickchang 法(Stratagene社) に基づいています。
挿入したいPCR断片に、挿入位置のベクター配列24 bpほどをつけておくと、これがメガプライマーとして環状のdestionation vectorにアニールし2段階目のPCRが起こります。PCR反応が終わった後、もともとの (メチル化されている) destionation vectorを制限酵素DpnIで消化し、形質転換すれば、PCR反応の結果できた新生コンストラクトを得ることができます。
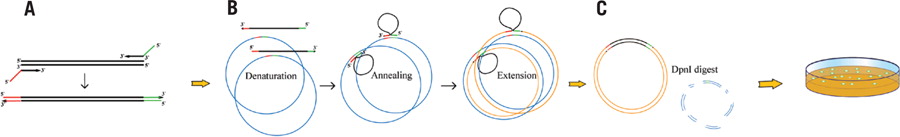
Source: https://www.future-science.com/doi/10.2144/000113418?url_ver=Z39.88-2003&rfr_id=ori:rid:crossref.org&rfr_dat=cr_pub%3dwww.ncbi.nlm.nih.gov
これを改良したのがTransfer-PCR (TPCR) システムで、別々だった断片増幅と挿入反応を、ドナーベクター、destination ベクター、プライマーを1つのPCR反応にまとめました (Transfer-PCR (TPCR): a highway for DNA cloning and protein engineering. J. Struct. Biol. 2011)。
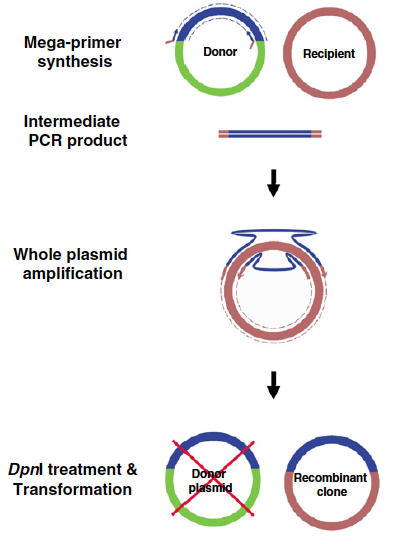
Source: https://www.sciencedirect.com/science/article/pii/S1047847711001109?via%3Dihub
Circular Polymerase Extension Cloning (CPEC) は、リニアにしたベクターにPCR断片を挿入するので、DpnI処理が最後に不要になるという手法です (Circular polymerase extension cloning of complex gene libraries and pathways. PLoS ONE 2009)。
PCRによるクローニング法は、最終的なコンストラクトは線形な増幅によって得られることになります。もし収量の少なさが問題ならexponential mega-priming (EMP) クローニングが有効かもしれません。これはPCR産物の一端のみに20-25 bpほどの相補的な配列を用意しておき、最終産物を指数的に増幅する方法です (Exponential megapriming PCR (EMP) cloning–seamless DNA insertion into any target plasmid without sequence constraints. PLoS ONE 2012)。
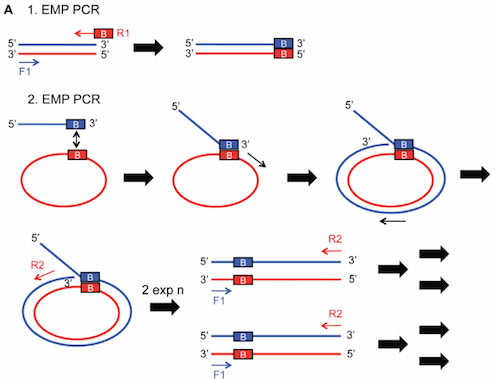
Source: https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0053360
Ligase cycling reaction(LCR)では、一本鎖のbridging DNAオリゴヌクレオチドを多数のPCR産物を連結するために使います (Rapid and reliable DNA assembly via ligase cycling reaction. ACS Synth. Biol. 2014)。
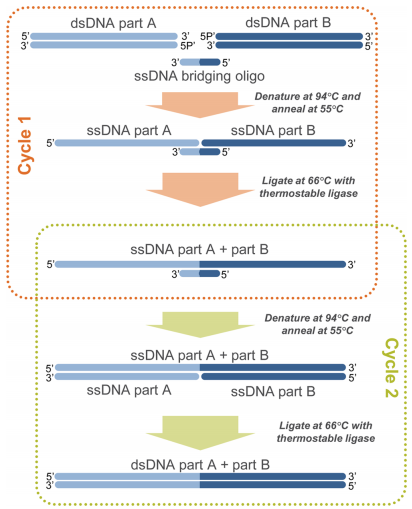
Source: https://pubs.acs.org/doi/10.1021/sb4001992
ブリッジさせるオリゴは2つのパーツに相補的なものをつなげて作りますが、それぞれTm 60℃を目安にするとのことです。耐熱性ligaseの条件 (66℃)、加えるパーツやブリッジオリゴの濃度 (3, 30 nM) などいろいろ検討して最適化された結果が論文に記載されています。
Gibsonアセンブリよりも、特にパーツの数が多くなった時に有利であり、10パーツを超えるものも同時に連結できるそうです。
突然変異や欠失のあるコンストラクトの作成
QuickChange法(Agilent社)は、PCR反応を使って短いDNA領域 (50 bp未満) のいくつかの塩基を欠失させたり置換させる最も一般的な部位特異的突然変異誘発法です。配列の中央に導入したい変異を持ち両側には10~15塩基の相補的な領域を持つプライマーと、もともとの環状プラスミドDNAが必要で、プライマーは鋳型DNAにアニーリングします。
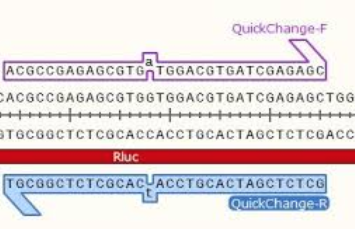
プライマー設計の例
アニーリングと伸長を繰り返すと、目的の変異を含むプラスミドDNA全体が増幅され、もともとの鋳型DNAはDpnI制限酵素により選択的にdigestionしてから形質転換するという方法です。
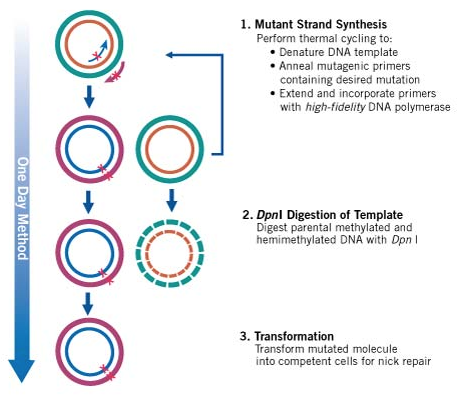
Source: https://www.chem-agilent.com/contents.php?id=300573
複数の部位に同時に突然変異を導入するには、QuikChange Multi Site-Directed Mutagenesis Kitを使用することもできます。この方法はQuickChange法と同じ原理ですが、ポリメラーゼに加えて (企業秘密の) 酵素ブレンドを加えることで、できた断片の間のニックを閉じるという手法です (Creating randomized amino acid libraries with the QuikChange Multi Site-Directed Mutagenesis Kit. Biotechniques 2002)。この方法は縮重プライマーを用いたランダム突然変異ライブラリーの作成にも適しています。
より大きなDNA断片を挿入したり置換する必要がある場合、制限酵素を使わないPCRベースのクローニング手法を使うことができます。
ある報告によれば、2.4 kbまでの欠失と3.5 kbまでの挿入がPCRベースのクローニング手法で導入されています。
タンパク質の共発現系
タンパクの共発現は大きく3つの方法があります。
1つ目として、それぞれのタンパクを作る複数のプラスミドを用意する方法。2つ目として、1つのベクターの中に、それぞれのタンパクがそれぞれ異なるプロモーター下にあるような発現ベクターを用意する方法。3つ目として、複数のタンパクが1つのmRNAとして転写され、その後IRESや2aペプチドのようなもので別々に翻訳されるという戦略です。
リコンビナーゼやPCRに基づく方法の多くは、複雑なコンストラクトを作るために組み合わせることができ、合成生物学の分野で広く使用されています (Bricks and blueprints: methods and standards for DNA assembly. Nat. Rev. Mol. Cell Biol., 2015)。
広範に利用可能でかつ自動化も可能なワークフローであるACEMBL技術は構造生物学の分野で広く利用されています (Recombinant expression and reconstitution of multiprotein complexes by the USER cloning method in the insect cell-baculovirus expression system. Methods 2016)。
酵母の相同組換えを利用して、複数のパーツを組み立てるという方法もあります (Recombination-mediated PCR-directed plasmid construction in vivo in yeast. Nucleic Acids Res. 1997)。線状化したプラスミドと、およそ30-40 bpのホモロジーを両端に持つインサートを酵母の中に導入するだけです。
植物におけるタンパク質発現のためのGolden Braid (GoldenBraid: an iterative cloning system for standardized assembly of reusable genetic modules. PLoS ONE 2011), USERec (USER friendly DNA recombination (USERec): gene library construction requiring minimal sequence homology. Methods Mol. Biol. 2014)、UNSアセンブリ (Unique nucleotide sequence-guided assembly of repetitive DNA parts for synthetic biology applications. Nat. Protoc., 2014)、およびadvanced Gibsonアセンブリ (A modular assembly platform for rapid generation of DNA constructs. Sci. Rep., 2016)など、多数の組換えに基づく技術が合成生物学の分野で開発されています。
クローニングに役立つリソース
NEB Tools & Resources: クローニングに関するツールやリソースをまとめたものです
RestrictionMapper: 配列を入れると、制限酵素認識部位を表示してくれるツール
NEBcutter: 配列を入れると、制限酵素認識部位を表示してくれるツール
NEBioCalculator: DNAクローニングで必要となるいろいろな情報の計算機です
Codon Adaptation Tool: コドンの最適化をしてくれるオンラインツール
GeneCards: 人の遺伝子についての網羅的なデータベース
Serial Cloner: クローニングに役立つソフトで、使い方の動画もあります
まとめ
最後に今回の内容をまとめます。
- クローニング方法は制限酵素以外にもたくさんある
- 中でもPCRベースのシームレスなクローニングが主流になりつつある
- 役立つリソースも有効活用
今日も【医学・生命科学・合成生物学のポータルサイト】生命医学をハックするをお読みいただきありがとうございました。