バイオインフォマティクス解析を行う時には、Linuxベースのコマンドを使うことが避けて通れません。
この記事ではLinuxコマンド入門について、これまでデータ解析に馴染みがない方に向けて紹介します。
この記事の内容
そもそもLinuxとは
WindowsやmacOSなどと同じく、LinuxというのはOS (オペレーティングシステム) の名前です。
商用のOSと異なり誰でも自由に使えるOSで広く普及し、ソースコードも公開されているということがブラックボックスを残さないという科学界の姿勢ともマッチしてバイオ系のデータ解析の標準になりました。
RNA-seqの解析に使うhisat2やsalmonなど、多くのツールはLinux上で動くように作られています。
Linuxは無料で環境構築できる上、すでにたくさんの良質な記事があるので、環境構築についてはこの記事では触れません。
macOSをお使いの方は、一部Linuxと異なる部分もありますが多くのコマンドはmacOSでも動かすことができ、ターミナルというアプリを起動してコマンドを入力することができます。
例えば、ターミナルに次のように入力しエンターキーを押してみてください。
cal
今月のカレンダーが表示され、今日の日付もマークされていますね。
このcalというのはカレンダー (calendar) を表示せよという命令になります。
このようにマウスで画面をクリックするのではなく、キーボードで命令 (コマンド) を出してコンピューターを操作することをCUI (Character User Interface) といいます。
画面のクリック操作を他の人に正しく伝えるのは困難ですが、コマンドは記録として残るため再現性が高く、科学界のツールの多くはCUIベースのものになっています。
ディレクトリの考え方
はじめてLinuxを使う場合、真っ先に把握したいのはディレクトリの概念です。
ディレクトリというのは、windowsやmacOSでいうところの「フォルダ」のことです。Linuxでは最上位のディレクトリが1つあり、全てのディレクトリはそこから階層構造になっていてたどることができます。
このうち、最上位の1つしかないディレクトリのことをルートディレクトリといい、現在自分がいるディレクトリーをカレントディレクトリ (ワーキングディレクトリとも) といいます。
カレントディレクトリを調べるのはpwdコマンドを使います。pwdはprint working directoryの略です。
pwdディレクトリの作成と移動
ディレクトリの移動にはcdコマンドを使います。cdはchange directoryの略です。
ここではdesktopに移動してみます。
cd desktop新しいディレクトリを作るためにはmkdirコマンドを使います。これはmake directoryということです。例えばtrainingという名前のディレクトリを作るためには
mkdir trainingと実行することで、デスクトップにtrainingディレクトリができます。今現在は先ほどのdesktopディレクトリにいますが、新しく作ったtrainingディレクトリの中に移動してみます。
cd trainingさらにここにdataというディレクトリを作ってみましょう。mkdirコマンドを使うのでしたね。
mkdir data作成したdataディレクトリの中に移動するには
cd dataでできますね。
それではここから1つ上のtrainingディレクトリに戻るにはどうすればいいでしょう? 1つ上の階層に移動するには ..を使います。
cd ..ということです。
このcd ..で1つ上に戻るというのはよく使うので把握しておきましょう。
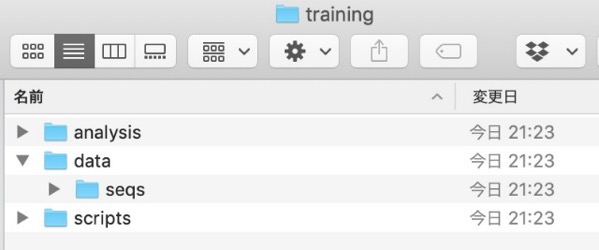
さらにこのdataディレクトリの中にseqディレクトリを作ってみます。さらに同時にscripts, analysisというディレクトリを作ってみます。
mkdir data/seqs scripts analysismkdirにはスペースをはさんで複数のものを渡すことができます。
すると、このようにtrainingディレクトリの中にanalysis, data, scriptsの3つのディレクトリがあり、さらにdataの中にはseqsディレクトリがあります。
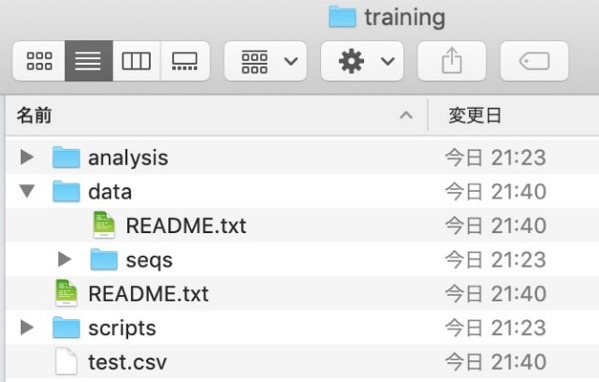
touchコマンドを使うと、空のファイルを作ることができます。例えば、現在のtrainingディレクトリにREADME.txtを、dataディレクトリの中にREADME.txtを作る場合には
touch README.txt data/README.txtテキストファイルだけでなく、例えばcsvファイルも作成することができます。
touch test.csv実際にファイルを確認すると、2つのテキストファイルと1つのcsvファイルが増えていることが分かります。
echoコマンドとブレース展開
次は文字の表示についてみていきます。一番基本になるのがechoコマンドで、これはその文字をターミナルに表示します。
例えば、Helloと表示したければ
echo 'Hello'とすればOKです。
{}で囲めば、それらを使った全てを表示します。少し分かりにくいので、p53, p63, p73を表示する例を見てみましょう。
echo p{5,6,7}3するとこのように、{}の中の文字が1つずつ使われ、順にp53, p63, p73と表示させることができました。
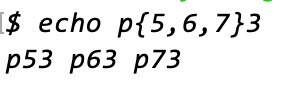
このように、{}内にコンマ区切りで書いた文字を展開して使うことを、ブレース展開 (brace expansion)といいます。
しかしこの方法では数が増えるとちょっと面倒ですね。例えばE3 ユビキチンリガーゼであるFBXW1から13まで全部表示するためには
echo FBXW{1,2,3,4,5,6,7,8,9,10,11,12,13}
と書かなければなりません。こういった場合には、最初と最後を..でつなぐ書き方が便利です。つまり今回の場合、
echo FBXW{1..13}とすれば、これら13個を同時に表記できます。
![]()
ブレース展開は複数組み合わせることができます。例えばFBXWシリーズだけでなく同じくE3リガーゼであるFBXLシリーズやFBXOシリーズも表記したいなら
echo FBX{W,L,O}{1..13}とブレース展開を2つ使うことで 3つのシリーズと各13個の遺伝子、合わせて39個をまとめて表示できます。
ブレース展開を応用したディレクトリ・ファイル作成とlsコマンド
ブレース展開は文字を表示するだけのものではありません。ファイルやディレクトリを作るためにも使われます。
例えば、read_1.fastqからread_4.fastqまで4つのfastqファイルを作ってみます。fastqファイルはRNA-seq等の次世代シークエンサーで得られる標準的なファイルです (今回はファイルの中身は空ですが)。
新しいファイルはtouchコマンドで作成することができるのでしたね。これとブレース展開を組み合わせれば、
touch read_{1..4}.fastqとすれば4つの新しいファイルがカレントディレクトリに作成されています。
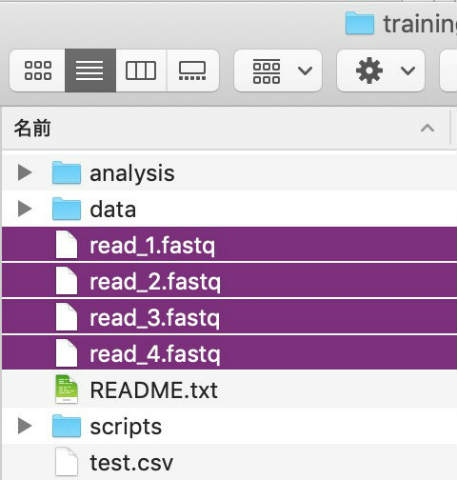
これまではファイルやディレクトリができたのかを1つ1つ「見て」来ましたが、この方法だとちょっと不便です。Linuxのコマンドラインでファイルの確認をするのがlsコマンドになります。
単純にlsコマンドを使うと、現在のカレントディレクトリにあるファイルやディレクトリが表示されます。
lsアスタリスク (*) を使えば、「どんなものでも」という意味を表現できます。例えば任意の文字のファイルで、最後が.fastqで終わるものを調べたければ
ls *.fastqとすればよく、4つのfastqファイルが出てきました。
また、カレントディレクトリ (training) にあるdataディレクトリの中にあるものを調べたければ
ls dataで確認できます。
現在のディレクトリから見たらすぐ下のディレクトリなのでこのように書けるわけで、こういった書き方を相対パスといいます。
一方で、絶対パスと呼ばれる、ルートディレクトリからの全ての階層を書いていく方法もあり、これはまた機会があるときに紹介します。
まとめに代えて
この記事では、Linuxコマンドでまず抑えておきたい6つのコマンド、つまり
pwd
mkdir
cd
touch
echo
ls
について紹介しました。特にtouch以外の5コマンドは非常によく出てくるので、重点的に復習をしてください。
この記事を出発点として、バイオ解析の基本を今後いろいろ紹介していきます。
次回の生命科学のためのLinuxシェルコマンドの基本1 【リダイレクトとオプション】では、画面ではなくファイルに出力する方法を中心にまとめています。
関連図書
この記事に関連した内容を紹介しているサイトや本はこちらです。
生命科学のためのLinuxシェルコマンドの基本1 【リダイレクトとオプション】
生命科学のためのLinuxシェルコマンドの基本2【パイプとコマンド置換】
今日も【生命医学をハックする】 (@biomedicalhacks) をお読みいただきありがとうございました。当サイトの記事をもとに加筆した月2回のニュースレターも好評配信中ですので、よろしければこちらも合わせてどうぞ