Biopythonはプログラミング言語Pythonをベースにした生命科学研究のためのパッケージで、その基礎的な使い方を3回に渡って紹介しています。
第1回目のBiopython入門 - 前編 【インストール ~ 簡単な使い方】ではDNAやタンパク配列の作り方を、第2回目のBiopython入門 - 中編 【NCBIデータベースやBlastへのアクセス】ではBiopythonを使ったデータベースやツールへのアクセスを紹介しました。
今回は、Biopythonでデータの可視化を行います。
系統樹データの可視化
生命科学のデータをPythonで可視化するためにはMatplotlibやSeabornといった汎用的なツールが論文等でよく使われていますが、Biopythonでは配列の表示と系統樹の図示がサポートされています。
Bio.Phyloパッケージは系統学的な情報を扱うためのモジュールで、この中に樹形図を描く機能が含まれています。
系統樹データを記述するファイル形式はいろいろありますが、その1つがNewick形式です。Newick形式はノードのペアを入れ子にした形で表現し、例えば
はAとBが、CとDがそれぞれつながり、その両者がさらにつながったものがさらにできるという大きなつながりと、E, F,Gの3つからなるつながりが、最終的につながって1つの系統樹になるということを意味しています。
このファイルをsimple.dndという名前にすると、Biopythonではこのように読み込んで系統樹を表示できます。
from Bio import Phylo
tree = Phylo.read("simple.dnd", "newick")
print (tree)
Phylo.draw_ascii(tree)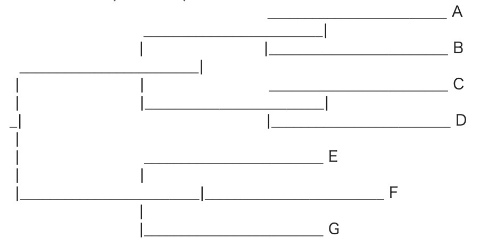
Phylo.draw(tree)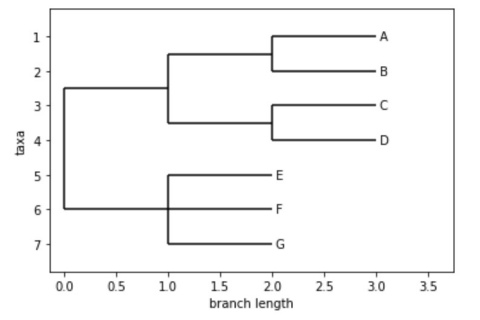
ゲノムデータの可視化
Bio.Graphics.GenomeDiagramというライブラリを使うと、ゲノム配列の表示をすることができます。今回はNC_005816.gbファイルを読み込んでみます (NCBI Genbankのページ、右上の小さい「Send To」> 「File」から取得できます)。
Bio.Graphics.GenomeDiagramは内部でreportlabというパッケージを使っているので、まずはreportlabというパッケージをインストールします (pipで入れられます)。
pip install reportlabまずはGenBankデータNC_005816.gbをBio.SeqIOで読み込み、そのrecordのうちfeatureが「gene」となっているものだけを取り出します。
from reportlab.lib import colors
from reportlab.lib.units import cm
from Bio.Graphics import GenomeDiagram
from Bio import SeqIO
record = SeqIO.read("NC_005816.gb", "genbank")
gd_diagram = GenomeDiagram.Diagram("Yersinia pestis biovar Microtus plasmid pPCP1")
# レベル1のフィーチャートラックを作る
gd_track_for_features = gd_diagram.new_track(1, name="Annotated Features")
# フィーチャートラックに新しいフィーチャーセットを作る
gd_feature_set = gd_track_for_features.new_set()
for feature in record.features:
if feature.type != 'gene': # geneタイプだけを取り出す
continue
if len (gd_feature_set) %2 ==0: # 偶数奇数で色を区別する
color = colors.blue
else:
color = colors.lightblue
# フィーチャーを追加
gd_feature_set.add_feature (feature, color= color, label=True)
# 線状に描く
gd_diagram.draw (format='linear', orientation='landscape', pagesize = 'A4', fragments = 4, start=0, end = len (record))
gd_diagram.write ('linear.png', 'PNG')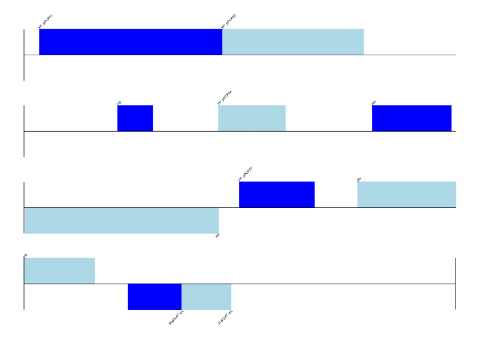
環状で描きたい場合には最後のところをこのように変更します。
# 環状に描く
gd_diagram.draw (format='circular', circular = True, pagesize = (20*cm, 20*cm), start=0, end = len (record), circle_core = 0.7)
gd_diagram.write ('circular.png', 'PNG')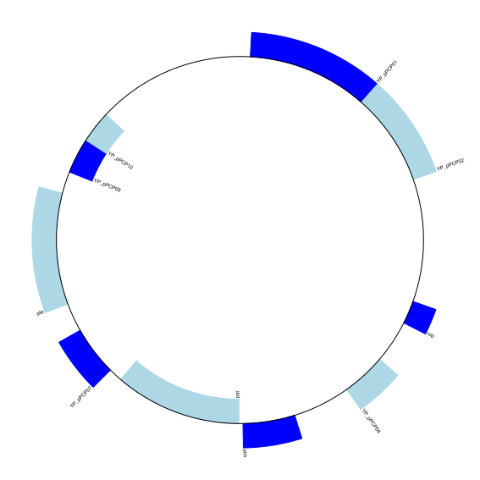
GenomeDiagramライブラリーでは、単に表示するだけではなく、フィーチャーに注釈を加えたり遺伝子部分を矢印にしたりすることが可能です。
from reportlab.lib import colors
from reportlab.lib.units import cm
from Bio.Graphics import GenomeDiagram
from Bio import SeqIO
from Bio.SeqFeature import SeqFeature, FeatureLocation
record = SeqIO.read("NC_005816.gb", "genbank")
gd_diagram = GenomeDiagram.Diagram(record.id)
gd_track_for_features = gd_diagram.new_track (1, name = 'Annotated Features')
gd_feature_set = gd_track_for_features.new_set ()
for feature in record.features:
if feature.type != 'gene': # geneタイプだけを取り出す
continue
if len (gd_feature_set) %2 ==0: # 偶数奇数で色を区別する
color = colors.blue
else:
color = colors.lightblue
# フィーチャーを追加
gd_feature_set.add_feature (feature, sigil ='ARROW', # 遺伝子を意味する矢印を追加
color= color, label=True, # 遺伝子名のラベルをつける
label_size = 12, label_angle=0)
# 4種類の制限酵素サイトをマークする
for site, name, color in [("GAATTC", "EcoRI", colors.green),
("CCCGGG", "SmaI", colors.orange),
("AAGCTT", "HindIII", colors.red),
("GGATCC", "BamHI", colors.purple)]:
index = 0
while True:
index = record.seq.find (site, start = index) # 配列上で制限酵素位置を検索
if index == -1 : break # もしなければ何もしない
# 位置の情報を作成
feature = SeqFeature (FeatureLocation (index, index + len (site)))
gd_feature_set.add_feature (feature, color=color, name=name,
label=True, label_size=10, label_color=color)
index += len (site)
# 環状に描く
gd_diagram.draw (format='circular', circular = True, pagesize = (20*cm, 20*cm), start=0, end = len (record), circle_core = 0.5)
gd_diagram.write ('circular_2.png', 'PNG')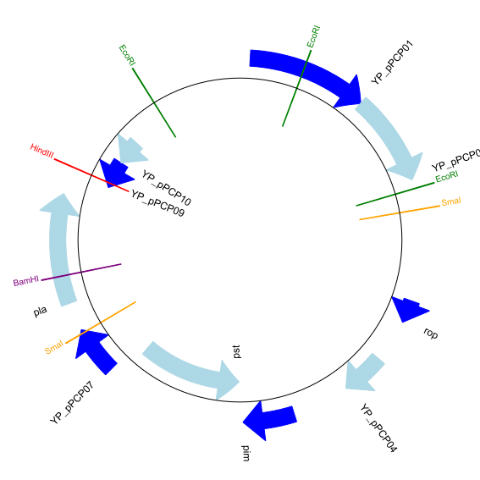
まとめに代えて
この記事では生命科学研究に不可欠な可視化について扱いました。冒頭にも述べたように可視化の多くはBiopython以外のツールで行われていますが、Biopythonにもこういったことができるということは知っておいても損はないと思います。
ちなみに、最後の配列情報の可視化にはSnapGeneをはじめ使いやすい専用のツールが色々あります。プラスミド管理ソフトウェア8選 【プラスミドの図の作成もできる】にもまとめているので合わせてご覧ください。
関連図書
この記事に関連した内容を紹介している本やサイトはこちらです。
Biopython入門 - 前編 【インストール ~ 簡単な使い方】
Biopython入門 - 中編 【NCBIデータベースやBlastへのアクセス】
プラスミド管理ソフトウェア8選 【プラスミドの図の作成もできる】
今日も【生命医学をハックする】 (@biomedicalhacks) をお読みいただきありがとうございました。
当サイトの記事をもとに加筆した月2回のニュースレターも好評配信中ですので、よろしければこちらも合わせてどうぞ