ある塩基配列がゲノム中にあるのか、あるならばどこにあるのかを知りたいことは多々あります。制限酵素サイトやCRISPRのガイドRNAなど、実験をする上で情報が必要なこともあります。
この記事では、そんな時に役に立つウェブツールGGGenome (ゲゲゲノム) を紹介します。
ゲノムの高速検索
GGGenome (ゲゲゲノム) は塩基配列を検索できるサービスで、300以上の生物ゲノムや転写産物 (RefSeqだけでなくmiRBaseなども含む) などに対応しています。
塩基配列を検索できるサービスは、他にもBLASTやBLATなどがありますが、これらはいずれも10塩基以上の配列でないと検索できません。
GGGenomeは6塩基以上であれば検索でき、しかもsuffix array (サフィックスアレイ) という技術を使って高速に検索できるのが特徴です。
まず、検索したい配列と、どのデータベースに対して検索するのかを選びます。
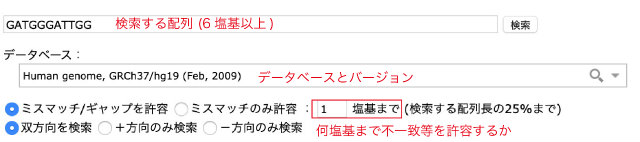
また、ミスマッチやギャップなどの不一致を何塩基まで許すのか (検索した配列と全く同じものを調べたいなら0塩基の不一致を許すという意味で0を入力) 指定します。
検索を実行すると、このように高速に結果が返ってきます。それぞれはUCSCゲノムブラウザにリンクしています。
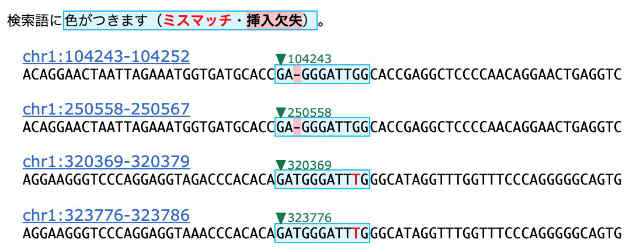
一番下にスクロールすると、結果をダウンロードできます。

UCSCSゲノムブラウザで結果を可視化する転写産物の高速検索
次に、UCSCゲノムブラザを使って、結果を可視化する方法を紹介します。
先程の画面、BEDファイルを指定して、出てきたページのURLをコピーしておきます。
続いてUCSCゲノムブラザを開き、My DataからCustom Tracksをクリックします。
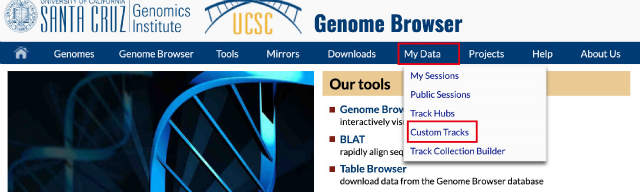
コピー済みのBEDファイルのアドレスを貼り付け、ゲノムバージョンをGGGenomeと同じものを選択してsubmitをクリックします。
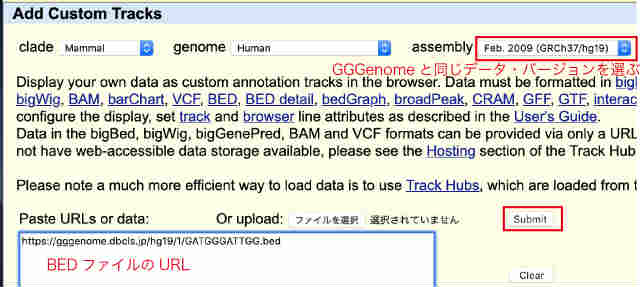
今回の場合は、1塩基のミスマッチを許しているので、9万箇所の該当する領域が見つかりました。
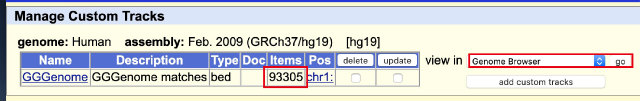
右にあるGenome Browserで可視化ボタンを押すことで、ブラウザ上段、GGGenomeというトラックに、ヒットした場所を表示することができます (黒いバーがヒットしたところです)。
![]()
ゲノムスケールを拡大縮小したり、目的の領域を検索するなどして結果をいろいろ見てみるといいでしょう。
転写産物の高速検索
ゲノムだけではなく、転写産物に対しても検索することができます。
GGGenomeのデータベースには、代表的な転写産物データベースであるRefSeqもあるので、それを選択して配列を検索するだけです。
あるいは、転写産物専用の高速検索ツールであるGGRNA (ググルナ) を使うという方法もあります。
GGGenomeと同じようにして転写産物を対象とした検索ができます。
関連サイト・図書
この記事に関連した内容を紹介しているサイトや本はこちらです。
https://youtu.be/TsF3Y8yEv3k
まとめ
最後に今回の内容をまとめます。
- ゲノムへの高速検索はGGGenomeが便利
- 結果をゲノムブラウザで簡単に可視化できる
- RNAバージョンのGGRNAもある
今日も【医学・生命科学・合成生物学のポータルサイト】生命医学をハックするをお読みいただきありがとうございました。