これまでの生命科学研究では、技術的な制約から多数の細胞を「すりつぶした」解析しかできませんでした。
近年の技術進歩により、1細胞 (シングルセル)レベルの解像度が出せるようになってきました。
この記事では、シングルセルレベルのゲノム、トランスクリプトーム、プロテオーム解析プラットフォームのさわりを紹介します。
シングルセルゲノム解析
1細胞全ゲノム配列決定のために、ゲノムは配列決定の前に増幅しなければいけません。
原理的にはPCRを用いた全ゲノム増幅法(Whole genome amplification from a single cell: implications for genetic analysis. PNAS 1992)で増幅することができますが、PCR法では偏りが生じやすいというのが問題です。
これに代わる方法として、φ29DNAポリメラーゼを使ったランダムプライミングによりゲノム全体を増幅するMDA(multiple displacement amplification)法 (Multiple displacement amplification法(Rapid amplification of plasmid and phage DNA using Phi29 DNA polymerase and multiply-primed rolling circle amplification. Genome Res. 2001)が開発され、広く使われています。
MDA法により全ゲノム増幅を行うことはできますが、依然としてバイアスが入ってしまうという問題がありました。
そこで、 multiple annealing and looping-based amplification cycles (MALBAC) と呼ばれる全ゲノム増幅アプローチが登場しました(Genome-wide detection of single-nucleotide and copy-number variations of a single human cell. Science 2012)。
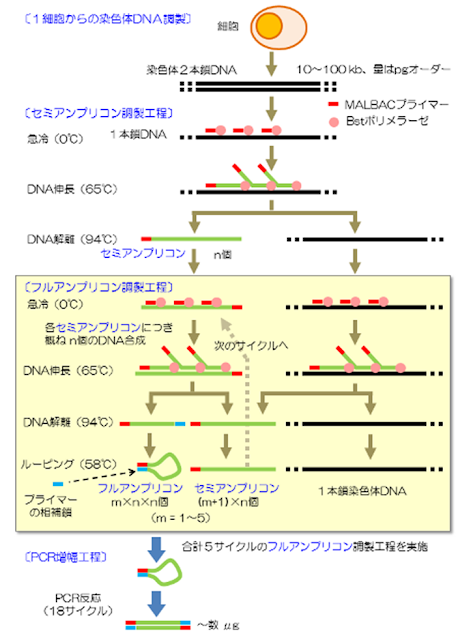
http://omics-club.blogspot.com/2013/04/20130409.html
MALBACに使うのはランダムオリゴと専用の配列をつなげたキメラプライマーで、最初のPCRにより3'末端および5'末端に用意した専用の配列同士が作る環状のアンプリコンを利用して、PCRのように指数乗ではなく線形にゲノム増幅でき、増幅に伴うバイアスを少なくできるという方法です。
目的とするゲノム領域を濃縮して配列決定する方法もたくさん開発されています。例えば、エキソームシークエンシングを1細胞解析に拡張した方法などが報告されています(Single-cell exome sequencing and monoclonal evolution of a JAK2-negative myeloproliferative neoplasm. Cell 2012)。
シングルセルトランスクリプトーム解析
シングルセルRNA-seqの主な技術的課題は、PCRによるバイアス補正と絶対定量ができる方法を開発することでした。この目標は、マイクロ技術(Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell 2015)と分子バーコード技術(Quantitative single-cell RNA-seq with unique molecular identifiers. Nat. Methods 2014)、さらにはいろいろなマイクロ流路プラットフォームの併用によって達成されています。
例えば、CytoSeqという方法は、20 pl程度のマイクロウェルへ希釈した細胞を加えてシングルセルにして解析する手法です(Combinatorial labeling of single cells for gene expression cytometry. Science 2015)。
各マイクロウェルには20 μm 程度の小さい磁気ビーズが入っていて、このビーズには、ユニバーサルPCRプライミング部位、細胞バーコード、unique molecular index (UMI)とそれぞれのmRNA捕獲配列を持つたくさんのオリゴヌクレオチドプライマーがついています。
UMIとは、例えば、黒、青、緑の3種類のmRNAがあるとしてそれらをシングルセルRNA-seq解析で定量する場合、ライブラリ調製の段階でそれぞれのmRNAに丸で示すUMIをつけておきます。PCRで増幅して量的な関係が崩れてしまっても、シークエンスでUMIが何種類あるかを調べれば、もともとその分子が何コピーあったかを推測することができます。
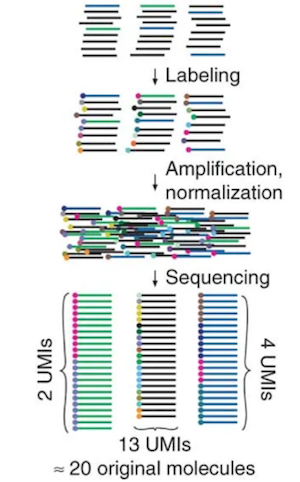
https://www.nature.com/articles/nmeth.1778
ウェルベースではなくマイクロ液滴ベースの方法もあります。例えばDropSeq法(Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell 2015)やinDrop(Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell 2015)などです。
この領域は非常に進歩が早いので、機会があればまた別の記事にまとめます。
シングルセルプロテオミクス解析
シングルセルプロテオミクスは、 マイクロ流体プラットフォーム、フローサイトメトリー、質量サイトメトリー (CyTOFなど) (Single-cell mass cytometry of differential immune and drug responses across a human hematopoietic continuum. Science 2011)のいずれかをベースにした方法がこれまで開発されてきました。
マイクロ流体プラットフォームを使った応用例では、microengraving技術(microengraving method for rapid selection of single cells producing antigen specific antibodies. Nat. Biotechnol. 2006)が最初に開発されたものです。
何千ものウェルがあるマイクロチップに希釈した細胞懸濁液を乗せると、ウェルの中には関心対象の細胞が全く入っていない場合もあるし複数入っている場合もある状態になります。調べたいタンパクに対する抗体がついているカバーガラスをマイクロチップ上に置き、いくつかの特異的な分泌タンパク質を捕捉することができます。
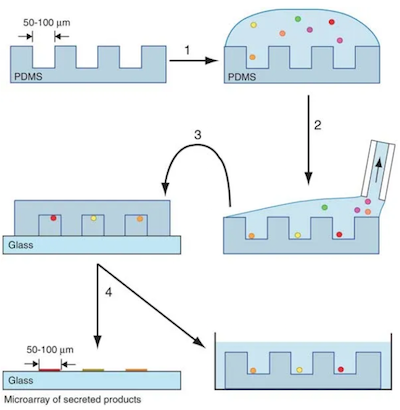
https://www.nature.com/articles/nbt1210
捕獲されたタンパク質を蛍光二次抗体で検出すれば、目的の抗体を分泌する細胞を同定し単離することも可能です。
これを大規模に行う、つまりカバースライドに多数の抗体がついているのがシングルセルバーコードチップ (SCBC)と呼ばれる方法です(Single cell proteomic chip for profiling intracellular signaling pathways in single tumor cells. PNAS 2012)。
シングルセルウェスタンブロット (scWesterns) という方法もあります(Single-cell western blotting. Nat. Methods 2014)。
scWesternは、0.5 mmくらいの非常に短い、光で活性化されるゲルに開けられた穴の中に細胞を入れてそこで細胞を溶かし、泳動した後に目的のタンパクに対する抗体で検出する方法です。
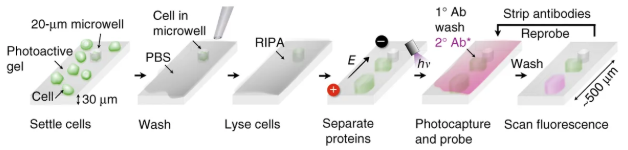
https://www.nature.com/articles/nmeth.2992
質量サイトメトリーは、遷移金属を含むタグがついた抗体で目的のタンパク質を標識し、1細胞にした後、質量分析装置で測定する方法です(Single cell profiling of potentiated phospho-protein networks in cancer cells. Cell 2004).
その他
シングルセルに関する技術は急速に進歩していて、分子レベルで単一細胞を包括的に分析できるいろいろな技術があります。
例えば、ある程度多数(>40)の分泌タンパク質(Highly multiplexed profiling of single-cell effector functions reveals deep functional heterogeneity in response to pathogenic ligands. PNAS 2015)や細胞表面マーカー (Single-cell mass cytometry of differential immune and drug responses across a human hematopoietic continuum. Science 2011)、およびリン酸化タンパク質(Single cell proteomic chip for profiling intracellular signaling pathways in single tumor cells. PNAS 2012)を同時にアッセイすることができます。
比較的最近開発された組織染色法である、連続ハイブリダイゼーションによるin situ RNAプロファイリング(Single-cell in situ RNA profiling by sequential hybridization. Nat. Methods 2014)を使えば、固定組織でのシングルセル解析を従来の免疫組織化学染色を大きく超えるスループットでできます。
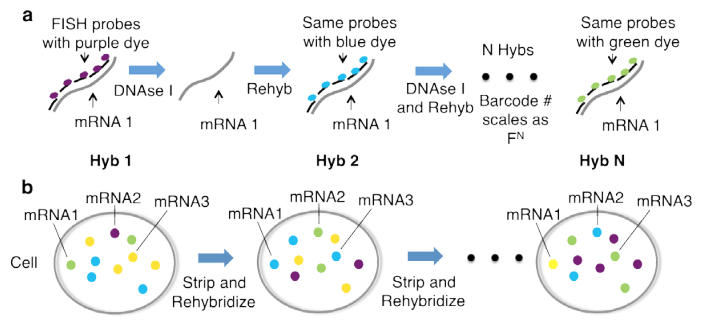
https://www.nature.com/articles/nmeth.2892#MOESM341
これは1つのmRNAに対して同じ色の蛍光標識をした多数のプローブでFISHを行い、DNase処理をしてプローブを分解し、次にまた別の蛍光標識をしたプローブでそのmRNAを検出して、というのを繰り返すことで、連続的に写真をとるとそのmRNA分子はある決まった色の順番 (紫→青→..) で出てきます。
多数のmRNAに対して、この色の並び順版を変えることで、それぞれの分子を画像からコンピューターで識別できるという仕組みです。
まとめ
最後に今回の内容をまとめます。
- シングルセル解析はゲノム増幅のバイアスを防ぐのが大事
- シングルセルプロテオミクスもある
- シングルセル技術は進歩が著しい
今日も【医学・生命科学・合成生物学のポータルサイト】生命医学をハックするをお読みいただきありがとうございました。