タンパク質の立体構造予測法
タンパク質の立体構造をアミノ酸配列だけから予測することは今も解かれていない難題ですが、似たようなアミノ酸配列を持つタンパクの構造がすでに分かっていれば、それを鋳型にして未知のタンパク質の構造をそれなりに高い精度で推察することができます。
このように似たようなタンパク質の構造を使う立体構造予測法をホモロジーモデリングといい、ホモロジーモデリングを行うツールの代表がSWISS-MODELです。
この記事ではSWISS-MODELの使い方を実際の解析例とともに紹介します。
SWISS-MODELのドキュメント
まずはトップページにアクセスします。SWISS-MODELにはいろいろなドキュメントが充実していて、それらには例えばトップページの右上からSWISS-MODELのドキュメントにリンクしています。
この記事では、例としてショウジョウバエのsuperoxide dismutase (SOD) についてホモロジーモデリングを行います (これはドキュメントにも記載されている例の1つです)。
タンパクに関しての事前知識は立体構造予測に非常に有用です。
今回のケースでは、SODの機能がラジカルを壊すことで、生理的にはホモダイマーを形成しており、1つのサブユニットには2つの金属 (CuとZn) 結合部位があることがこれまでの先行研究から分かっているということを利用してモデリングしていきます。
Templateの検索
新しいプロジェクトを開始するには、「Start Modeling」ボタンを押します。SWISS-MDELに入力できるのは4種類で、右端にその4つのボタンがあります。
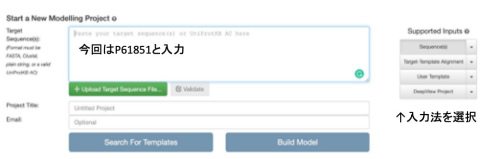
タンパクのアミノ酸配列やUniProtのIDを入力するだけでなく、Target-Template Alignmentオプションや、(鋳型となる構造を手元に持っている場合には)User Templateオプションも選択できます。DeepView というプログラムのユーザーならば、DeepView Projectオプションも用意されています。
今回はUniProtのアクセッションコードP61851を使ってみます。緑のボタンの右にあるものをクリックすると、適切なプロジェクトタイトルが自動で入力されるので便利です。

ホモロジーモデリングの最初のステップは、良いテンプレートを探すことで、これを行うにはSearch for Templatesボタンを押します。これによりBlastとHH Blitzが実行され、適切なテンプレートが提案されます。テンプレートサーチにかかる時間は10分程度で、ページをブックマークしておけばいったんこのページから離れても結果のページにアクセス可能です。
Templateの評価
Template searchが終わると、このような画面になり、検索したタンパクに対するtemplateのリストが表示されています。このリストはTemplate scoreが高い順に並んでおり、よりよいテンプレートと判断されるものがより上位にあります。
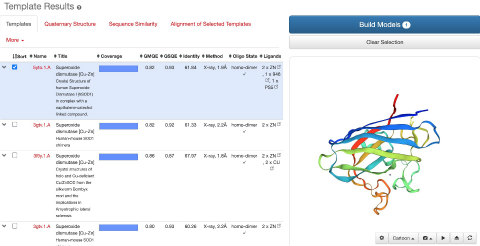
左端にあるボックスのチェックを付けたり外したりすることでそのテンプレートを使用するかどうかを選択可能です。選択したものは右側の3D viewerに表示されています。
2番目・3番目の列はSwiss-model template libraryにあるtemplateの名前です。4番目の列はCoverageで、今回検索したタンパク配列のうちどれくらいがそのtemplateでカバーされているかということ。今回のケースでは、配列は完全にカバーされています。
5番目のGMQEはGlobal Model Quality Estimateの略で、0から1の間をとる数字です。この値が1に近いほど、よいです。名前の通り、アラインメントの結果から推定したモデルの質を推定した値です。
6番目のQSQEはQuaternary structure quality estimateの略で0から1の値をとり、どれくらいよくinterchain相互作用があるのかを示しています。経験的にこの値が0.7を超えてくると良いモデルということができます。
7番目の列のIdentityはどれくらいのアミノ酸が正確にアラインメントされたのか。8番目の列「Methods」はその名前の通り。
9番目の列「Oligomeric State」もとても重要。今回はホモダイマーを作るという知見を反映しています。
10番目、最後の列は「Ligands」で、テンプレート中のリガンドを示しています。今回はCuとZnを含むという情報を利用することができます。
左端にある矢印をクリックすることで、さらに詳細を観察することもできます。
今回のtemplate候補の中で、上位にあって、既知の知見通りZnとCuをリガンドとして含んでいる上から3番目と6番目のモデルを選択してみます。
さらに、タブを切り替え、Quaternary structure tabをクリックすると、テンプレートがtree形式で表示されています。
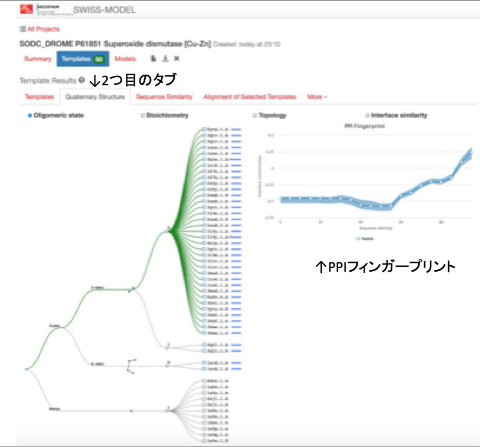
これをみると、まず、テンプレートはmonomerとsとHomomersに分かれています。Homomerの中はさらにdimersとtetramersに分かれています。dimerの中はさらにinterface similarity (どのようにタンパクchainがお互いにinteractしているか) によって2つにクラスター分けされているようです。
選択したモデルがこのツリーのどのあたりにいるのかも閲覧できます。モデルにマウスを合わせれば、それぞれのQSQEが表示されます。
PPI (Protein-Protein Interaction) Fingerprintは、x軸が配列のidentityでy軸がinterfaceの保存性をプロットしたものです。y軸の値が0より小さいと、interfaceに関わるアミノ酸がタンパク表面にあるアミノ酸より変異しにくいことを意味しています。
これはさまざまなカットオプによるモデル間のマルチプルアラインメントの結果から計算されています。
また、4つ目のタブであるAlignment of Selected Templatesでは、検索したアミノ酸配列と選択したtemplateについてのアラインメントが表示されています。
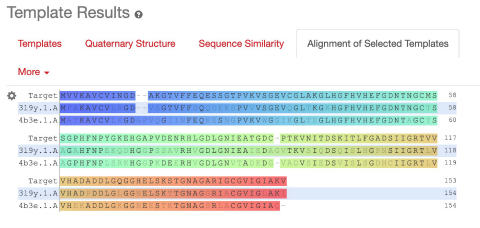
端にあるギアアイコンをクリックすると、DSSPやPSIPRED, SSproといった異なる2次構造予測プログラムを使い分けることも可能です。
立体構造モデリングとその評価
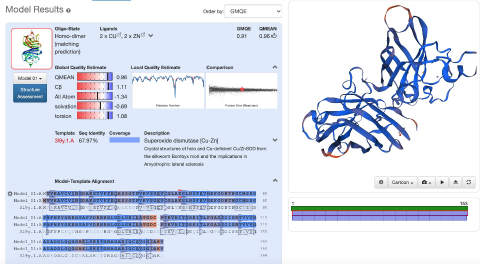
Templateを選択したら、あとはBuild Modelをクリックするだけです。少しすると、ホモロジーモデリングの結果が表示されます。
Swiss-modelではQMEANスコアをモデルの品質の見積もりとして使っています。
今回のモデルが、PDBにある他のアミノ酸配列・立体構造の関係と比較してどうかを調べるにはComparisonプロットを見ます。
中段の3番目に表示されていますし、クリックすると大きくすることもできます。
横軸はタンパクのサイズで、縦軸はQMEANスコア、個々の点はPDBデータベースの各モデルであり、スターが今回のモデルです。特に黒いところはZ-scoreが1未満のところになっています。
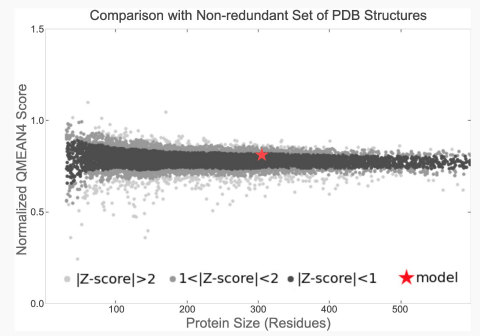
このケースでは、モデルは実験的に決定されPDBデータベースに登録されている構造と似ているということです。
QMEANスコアは中段の左端に表示されている4つの指標から計算されていて、QMEANスコアが-4を切ってくると、そのモデルはかなり信用できないということを意味します。
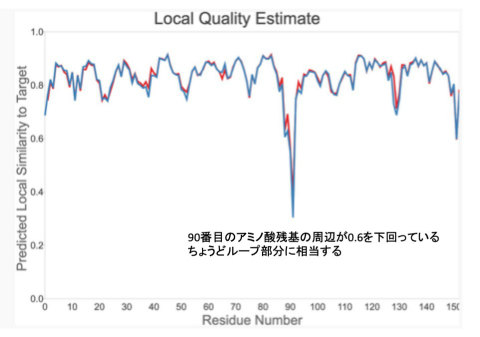
より重要なのは、モデルのどの部分がより信頼できて、モデルのどの部分があまり信用できないかを知ることです。それは中段の中央に表示されているlocal quality estimateをみると推察できます。
この値は0.6を切るとlow qualityであることが経験的に知られています。
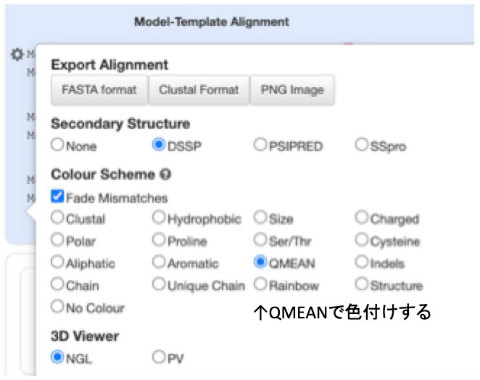
Model-Template Alignmentのところの左端にあるアイコンをクリックして、QMEANを選択すると信頼性が低いループの部分がオレンジで表示されます。
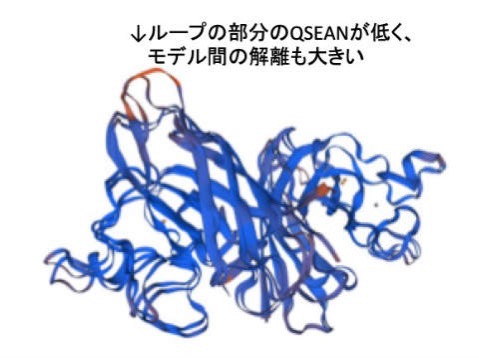
今回は2つのモデルができたので両方を選択してみると、青で表示されている部分は2つのモデルでほぼ一致しているが、ループ部分には差があることが分かります。
解析が終わったら、プロジェクト全体をこのボタンを押すことで自分のパソコンに保存することができます。

まとめに代えて
この記事では、SWISS-MODELの使い方の初歩を実際のスクリーンショットを使いながら解説しました。
ホモロジーモデリングは今なおタンパクの立体楮を高い信頼性で予測できる優れた手法です。構造が解かれていないタンパクを扱っている時にこそ、積極的に活用したいツールです。
関連図書
この記事に関連した内容を紹介している本はこちらです。
今日も【生命医学をハックする】 (@biomedicalhacks) をお読みいただきありがとうございました。当サイトの記事をもとに加筆した月2回のニュースレターも好評配信中ですので、よろしければこちらも合わせてどうぞ