TCGAは2006年から始まったプロジェクトで、いろいろながん種の遺伝子発現データ、遺伝子変異データなどを収集して公開しています。
そのTCGAデータに、特別なプログラミング教育を受けていなくても容易にアクセスできるようにしてくれるツールの1つがUCSC Xenaです。
この記事では、Xenaを使ったがん細胞の遺伝子発現・変異データを閲覧する方法を見ていきます。
この記事の内容
Xenaを使ったTCGAがんデータの表示
UCSC Xenaは、大規模な公開がんゲノムデータをブラウザ上で解析できるツールです。UCSCはゲノムブラウザでも有名ですね。
さまざまながんの遺伝子変異や構造バリアント、コピー数異常、遺伝子発現、エピジェネティック変化、ATAC-seqといった実験データと、実際のがん患者さんの表現型を自由に組み合わることができます。
まずはUCSC Xenaにアクセスします。 左側のところ「Help me select a study」を選択し、その下にLung Cancerと打ち込むと、該当するデータの候補がリストアップされます。 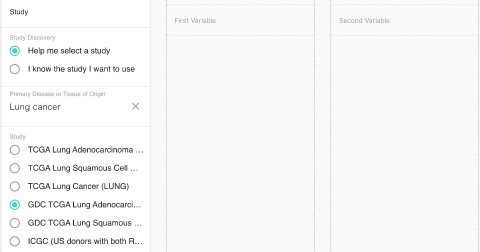
ここではGDC TCGA Lung Adenocarcinomaを選びました。TCGAは2006年から始まっていて、その間にレファレンスゲノムが新しくなりました。このGDCから始まるデータセットは、新しいバージョンのゲノムであるhg38にアラインメントさせたデータです。
ちなみにTCGAについてはがんゲノムのデータベースと解析ツール 【コマンド不要】でも紹介しています。
データを選んだら、一番下の「Done」をクリックし確定させます。
2列目以降には表示したい遺伝子を書いていきます。今回は分子標的薬の標的にもなるEGF受容体 (EGFR) について、遺伝子変異・コピー数異常・体細胞変異をチェックします。 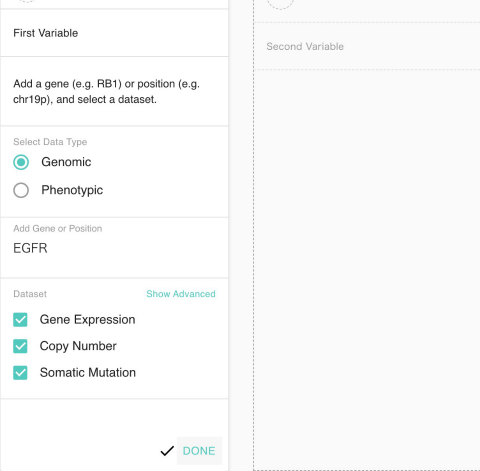
下側のDoneをクリックすると、このように簡単にヒートマップを書いてくれます。 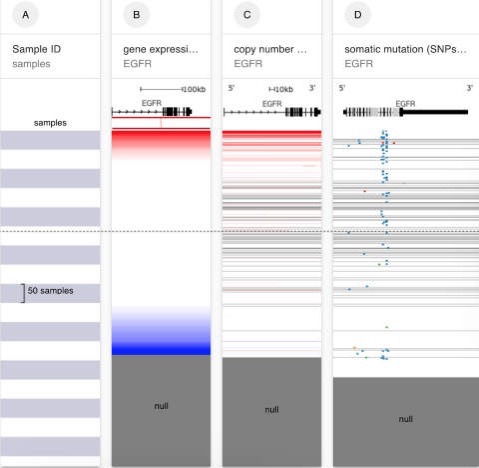
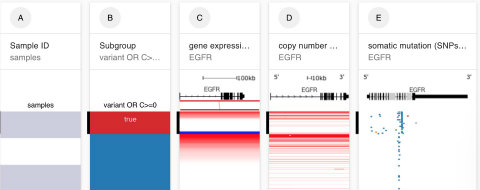
AのSample IDというカラムにも書かれていますが、青と白はそれぞれ50人の患者を示しており、それぞれの患者さんについて、B, C, DでそれぞれEGFRの遺伝子発現、コピー数異常、突然変異情報が書かれています。Bのカラムで優先してソートして表示されています。
現状では灰色、nullと書かれているそのデータがない患者さんも含んだプロットになっていますが、欠損データを除きたいなら一番上のところに「!=null」と入力し、その右にある逆向き三角形をクリックしてFilterをかければOKです。 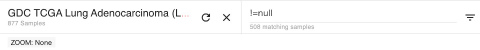
スペースは入れないように注意してください。
遺伝子発現と生存予後との関係を調べる
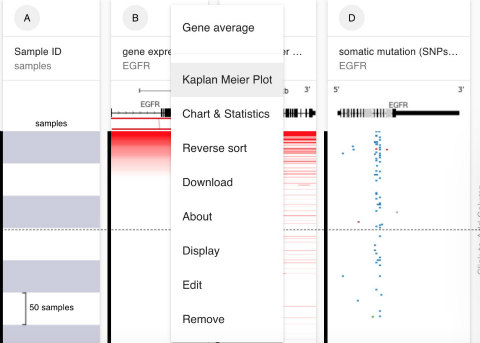
各列のところにカーソルを合わせ、黒い点をクリックしてみます (ここではB列のEGFR遺伝子発現のカラムについて見てみます)。

するといくつかの項目が現れ、これらの解析が簡単にできるようになります。試しにKaplan Meier Plotを選択してみましょう。
簡単にEGFR高発現/低発現群での生存曲線を比較することができます。今回の場合、p値から判断してEGFRの発現量については有意差はありません。
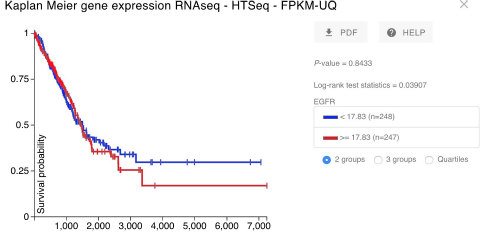
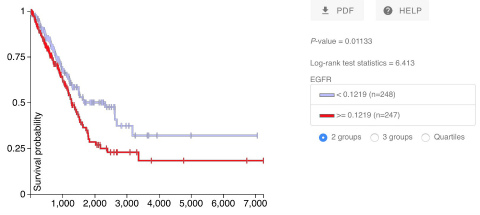
しかし隣のC列にあるコピー数異常について調べてみると、EGFRのコピー数が増加している患者さんでは予後不良と言えそうです。
カプランマイヤー生存曲線をRで解析する方法もRによる生存時間解析 【survivalパッケージでカプランマイヤー・コックス比例ハザード】で紹介しているので、Rに馴染みがある方はデータを自分のパソコンで解析することもできます。
遺伝子変異やコピー数異常と遺伝子発現との関係を調べる
EGFRの変異はよく調べられる項目です。今回見つけたコピー数増加と合わせて、EGFR遺伝子発現そのものには何か関係があるのでしょうか? まず上段にvariant OR C>=0と入力して「New Column」をクリックします。
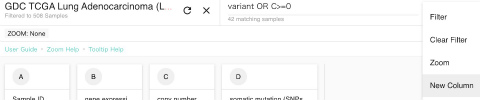
これは遺伝子変異がある (variant) か、またはコピー数異常を示すカラムCの値が0以上かを判定しなさいという意味で、カラムCはlog2表示なので実際にはコピー数が2倍以上になっているかどうかを判定します。
結果は新しくB列に作成され、条件を満たす (True) か否か (False)と返ってきます。
Bのところにある「Chart & Statistics」をクリックし、
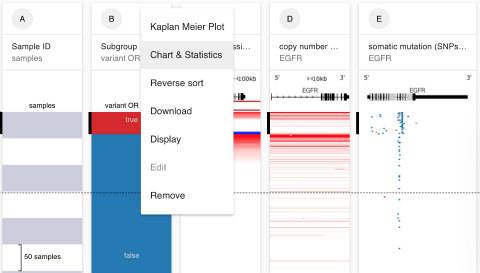
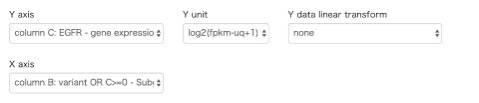
出てきた画面の下側にある「Y axis」を「Column C」(遺伝子発現), 「X axis」を「Column B」(コピー数2倍以上または遺伝子変異) を選択してみます。
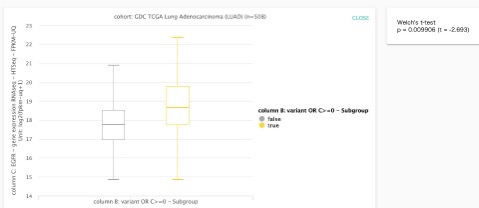
するとこのような画面になり、遺伝子変異やコピー数異常がある患者さんはEGFRの発現量も有意に (右上にp値が表示されています) 増加するということが分かります。
がんと正常の遺伝子発現比較を行う
Xenaにはがんだけでなく正常のデータも入っています。GTEx (Genotype-Tissue Expression) という、人の組織ごとの遺伝子発現を網羅的に調べる国際プロジェクトでデータが取得されています。
もう一度トップに戻り、GTExで検索してみます。TCGA TARGET GTExという3つのプロジェクトのデータが入ったデータセットを使います (ちなみにTARGETは小児がんについての国際プロジェクトです)。
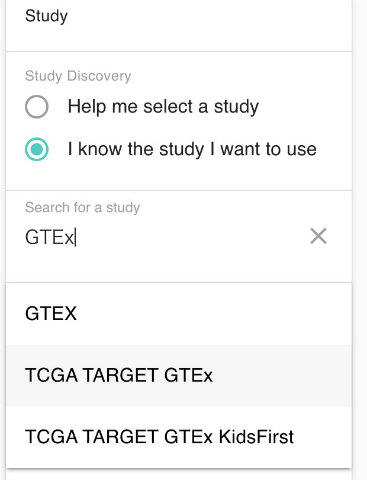
ここには19131サンプルが含まれますが、それぞれが何かについてはSelect Data Typeで「Phenotypic」、次の項目で「Study」を選んで「Done」すると表示されます。
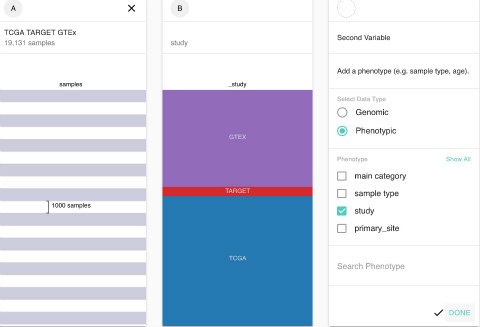
TARGETは少なく、ほとんどTCGA (がん) とGTEX(正常) だということが分かります。
今回はThe prognostic landscape of genes and infiltrating immune cells across human cancers (Nat. Med. 2015)という論文で報告された転写因子FOXM1の発現量について見てみます。この遺伝子はがんで高発現になることが知られています。
前と同様にやると、報告通りがんで赤くなっている (高発現) ことが分かります。 遺伝子はまとめて入力することもできます。例えばFOXM1が発現を制御しているPLK1やBIRC5, CCNB1をまとめて表示してみます。
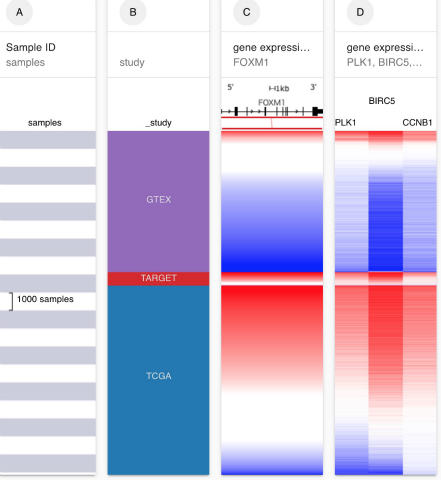
上流の転写因子の影響を受け、下流の因子も正常とがんでは大きく発現量が変動することが分かります。
この記事ではXenaの使い方の概略を紹介しました。バイオインフォになじみがなくても、あるいは極端な話、家からでもがん研究ができるということが分かっていただけたと思います。
関連図書
この記事に関連した内容を紹介している本はこちらです。
Rによる生存時間解析 【survivalパッケージでカプランマイヤー・コックス比例ハザード】
今日も【生命医学をハックする】 (@biomedicalhacks) をお読みいただきありがとうございました。
当サイトの記事をもとに加筆した月2回のニュースレターも好評配信中ですので、よろしければこちらも合わせてどうぞ