生命科学実験でおそらく初めてやることになるのが、プラスミドのコンストラクト構築とその抽出でしょう。生命科学系の論文で、プラスミドを全く使わないというものはごく少数です。
ある程度経験を積まれた方にはあたり前の内容ですが、この記事ではプラスミド抽出を初めて行う学生さんに向けて、簡単な原理とプロトコル、注意点をまとめます。
大きく3種類あるプラスミド抽出法
まず迅速ライゲーション・トランスフォーメーションの方法 【日常のDNAワークをより速く】で紹介したように、プラスミドを構築して大腸菌で増やします。
そのプラスミドの抽出方法は、大きく3種類あります。
1つは本記事で紹介するアルカリSDS法で、その名前の通り途中にアルカリ+SDS溶液を加えるステップがあります。
2つ目はボイルミニプレップ法で、その名前の通り途中でお湯に入れるステップがあります。ボイルミニプレップは1本のチューブでプラスミドがとれます。
そして3つ目は、市販のキットを使う方法です。この方法は実際には本記事で紹介するアルカリSDS法を使っているのですが、高価なカラムを通すことで、より「きれいな」プラスミドを取ることができます。
市販のキットにはミニ (Mini) プレップやミディ(Midi) プレップ、マキシ (Maxi)プレップといった名前がついていますが、これは対象となる大腸菌液の量を表していて、だいたいですがMiniだと~5 mL、Midiだと~50 mL、Maxiだと数百 mLの培養液を使うことが多いと思います。
どうしても市販のキットを使わないといけない場合もありますが、DNAの切り貼りやコンストラクトが正しくできているかのチェック、ラフなトランスフェクション実験ならこの記事で紹介するキットを使わない方法で十分です。
ボイルミニプレップについてはボイルミニプレップによるプラスミド抽出 【大量サンプルの時に便利】でまとめていますので、この記事では昔ながらのアルカリミニプレップに改良を加えた迅速ミニプレップについて紹介します。
用意するもの
アルカリミニプレップでは、次の3つの溶液が必要になります (他にTEなど一般的なDNAワークにおける試薬も必要です)
0.5M EDTA (pH8.0) 20 mL
Milli-Q water 930 mL
10% SDS 100 mL
Milli-Q water 880 mL
Milli-Q water ~800 mL
HCl で pH 5.8 に調整後、1000 mL にメスアップ
プロトコルと原理
プラスミドをトランスフォーメーション語、大腸菌を培養し、1.5 mLチューブに移す
↓
15000rpm, 1分遠心し、上清を吸引
↓
200 μlのp1を加え、しっかりとボルテックス
↓
200 μlのp2を加え、5回転倒混和
↓
すぐに200 μLのp3を入れ,チューブを5回転倒混和
↓
すぐに15000rpm, 4℃, 5分遠心。この間にもう1セットのチューブを用意し500 μlのイソプロプルアルコールを入れておく
↓
遠心後の上清をイソプロを入れておいたチューブに移す
↓
5回転倒混和した後,すぐに15000 rpm, 4℃で5分遠心
↓
上清を吸引, 1度吸引したら30秒静置して壁の液を落とした後,再び吸引しイソプロを完全に除く。
↓
70%エタノールを1mLずつ各チューブに入れる
↓
フタをして3回転倒混和,15000 rpm, 4℃で1分遠心
↓
ペレットを目で確認しながら,ゆっくりsupを捨てる
↓
軽くフラッシュ、エタノールを完全に除く
↓
100 μlのTE (RNaseが入ったもの) を入れ,撹拌溶解。
↓
一部を制限酵素処理し、電気泳動で確認。電気泳動はアガロースゲル電気泳動の方法と原理 【失敗原因の考察も】にまとめています。
吸光度で濃度が測定できないわけ
多くの場合、DNAの濃度は260 nmの吸光度を調べて計算しているでしょうし、260/280比の基本: DNA濃度を吸光度で測る 【原理と注意点】にも書きました。
しかしこの方法では実際の濃度よりも高く出てしまいます。
その理由は、この方法ではRNAも入ってきていて、RNase処理はしているものの、フリーのヌクレオチドがまだたくさんいる状態だからです。これらが260 nmを吸収しているのです。
そのため、実際の260 nmの吸光度はプラスミド量を反映しません。プラスミドの濃度を測定するには、電気泳動をして、マーカーの濃さと比較しましょう。流すマーカーの量を決めておけば、それをベースにして、どれくらいのDNAがそこにあるのかを見積もることができます。
フェノール処理不要のわけ
昔のプロトコル集には、この後は除タンパクを行うフェノール処理を追加するプロトコルがたくさん掲載されています。
なぜフェノール処理が必要だったのでしょうか?一番大きな目的は、大腸菌が本来持っているendonucleaseというDNaseの一種を除きたいからです。
せっかくプラスミドを抽出したのに、そこにDNaseがあったら時間経過とともに分解してしまいますね。だからこそフェノール処理をしていたのです。
実は使用する大腸菌に特徴があるのです。どんな大腸菌でもいいわけではありません。
大腸菌の中にはendonucleaseが欠損している株があります。よく使われているものだと、例えばJM109とかDH5αなどがあります。
endonucleaseをコードする遺伝子はendA1というのですが、目的の大腸菌株のgenotypeを調べ、そこにendA1が書かれていれば、endonucleaseが機能しておらず、したがってここで紹介したフェノールなしの迅速ミニプレップが使える大腸菌株ということになります。
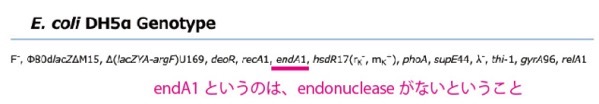
しっかりと大腸菌株を選べば、迅速にプラスミド抽出ができます。大腸菌の遺伝子型を調べることで他にもたくさんのことが分かるので、いつか記事としてまとめようと思います。
関連図書
この記事に関連した内容を紹介しているサイトや本はこちらです。
ボイルミニプレップによるプラスミド抽出 【大量サンプルの時に便利】
フェノール・クロロホルム抽出とDNA精製 【フェノクロ試薬の役割も】
迅速ライゲーション・トランスフォーメーションの方法 【日常のDNAワークをより速く】
今日も【生命医学をハックする】 (@biomedicalhacks) をお読みいただきありがとうございました。当サイトの記事をもとに加筆した月2回のニュースレターも好評配信中ですので、よろしければこちらも合わせてどうぞ