DBSCAN (Density-based Spatial Clustering of Applications with Noise) は非常に強力なクラスタリングアルゴリズムです。
この記事では、DBSCANをPythonで行う方法をプログラムコード付きで紹介し、DBSCANの長所と短所をデータサイエンスを勉強中の方に向けて解説します。
この記事の内容
DBSCANとは?
データを複数にクラスタリングする方法は、例えば階層的クラスタリングやK-means法などいろいろ知られていますが、DBSCANもクラスタリングを行うアルゴリズムの1つです。
大まかな仕組みとして、データの集合について、互いに密接にきっちり詰まっている点を同じグループにまとめ、低密度領域にある点を外れ値と判定します。
Density-based Spatial Clustering of Applications with Noise (DBSCAN)という名前のDensityはご存知の通り密度という意味なので、データの密度を利用してクラスタリングを行う方法なのです。
DBSCAN (日本語では密度準拠クラスタリングと呼ばれます)は、Pythonやいくつかのツールを使えば簡単に動かすことができます。
この記事の残りの部分では、ちょっとしたデータセットを使ってDBSCANのパラメータを調整してその使い方を見ていきます。
DBSCANとは?
まず、今回使ってみるデータセットを作ってみます。
データセットを作成する
データサイエンスでよく使われているPython言語で作られたscikit-learnというパッケージの中にあるmake blobs関数を使って作っていきます。
from sklearn.datasets import make_blobs
import seaborn as sns
import matplotlib.pyplot as plt
import numpy as np
centers = [[1, 0.5], [2, 2], [1, -1]] # 答えは3つのクラスターに分かれる
stds = [0.1, 0.4, 0.3] # それぞれのクラスターの標準偏差 (ばらつき具合)
X, labels_true = make_blobs(n_samples=1000, centers=centers, cluster_std=stds, random_state=0)
fig = plt.figure(figsize=(10, 10))
sns.scatterplot(X[:,0], X[:,1], hue=["cluster-{}".format(x) for x in labels_true])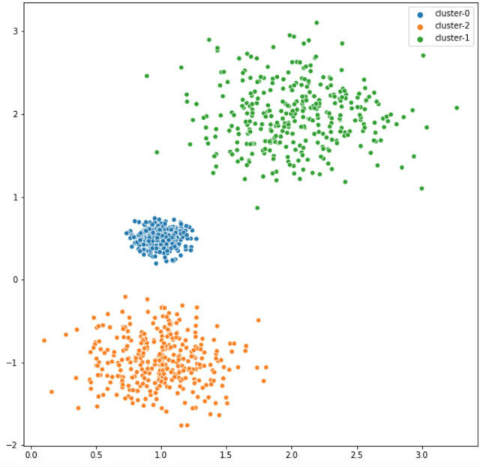
ここではご覧のように3つの郡に属するデータを作成しました。密度の異なるクラスターを作っているのは、クラスタリングを少し難しくするように、というデモ目的です。
DBSCANには2つの重要なパラメーターがある
DBSCANにはいくつかのパラメータがありますが、その中でも2つのパラメータが特に重要です。
min_points (min_samples)パラメータ
DBSCANのアルゴリズムを補足します。DBSCANでは、全てのデータ点はコア点(core points)、到達可能点、外れ値に分類されます。
この分類を決めるのが、min_points パラメーターで、簡単に言えばepsパラメーターで決まった半径の円の中にmin_pointsパラメーター以上の点が集まっていればコア点と判断します。
少し分かりにくいので具体例を見てみましょう。次の図を見てください (wikipediaから引用しています)。
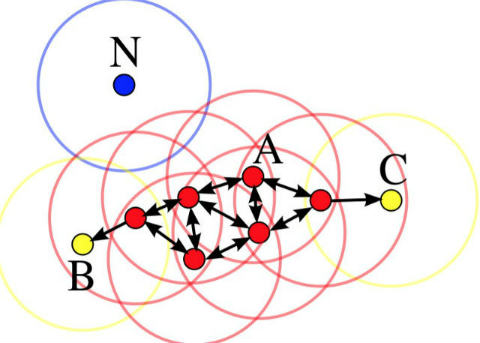
例えば、min_points=4だったとします。そうすると点Aやその他の赤色の点は「コア点」です。というのも、それぞれの円の中に自分自身も含めて少なくとも4つの点があるからです。
一方、BやCは4つの点がないのでコア点ではありません。しかし近くにある赤い点からeps半径の中に入っているので「到達可能点」と判断します。
コア点はそこから到達可能な全ての点とクラスターを作ります。今回の場合、A, B, Cで書かれた全ての点は1つの同じクラスターになります。
一方で点Nはどうでしょうか? この点は自身の円の中に1つしかないのでコア点ではなく、A/B/Cの点からも遠いので「到達可能点」でもありません。こういうものを「ノイズ点」といいます。
つまり今回の場合、N点はノイズ、それ以外は1つの同じクラスターと判断されるということです。
このようにDBSCANにおいてはepsとmin_pointsパラメーターが重要だということが直感的に理解できたと思います。
DBSCANをpython/scikit-learnで実装する
それではscikit-learnを使った実装です。幸いなことに、すでに用意されているクラスを呼び出し、自分のデータに当てはめるだけです。
コードの例はこちらです。
from sklearn.cluster import DBSCAN
db = DBSCAN(eps=0.5, min_samples=10).fit(X)
labels = db.labels_
fig = plt.figure(figsize=(10, 10))
sns.scatterplot(X[:,0], X[:,1], hue=["cluster-{}".format(x) for x in labels])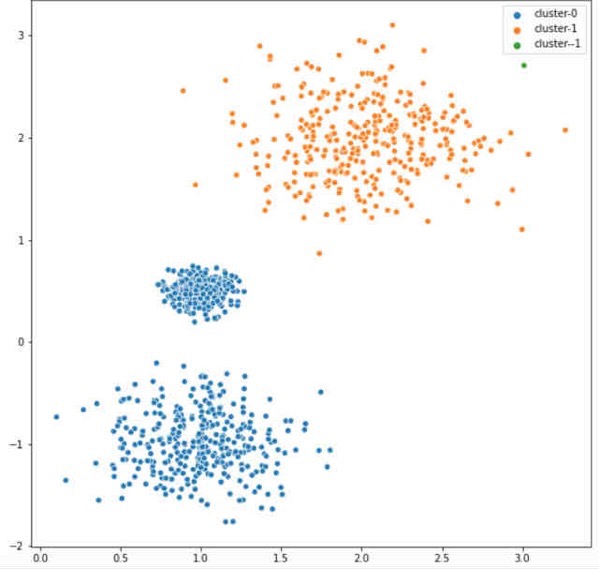
これをみると、3つではなく2つの大きなクラスターと判定されてしまいました。ここでハイパーパラメータepsを変化させてもう一度DBSCANクラスタリングしてみます。
import numpy as np
import seaborn as sns
import matplotlib.pyplot as plt
from sklearn.cluster import DBSCAN
fig = plt.figure(figsize=(20, 10))
fig.subplots_adjust(hspace=.5, wspace=.2)
i = 1
for x in range(10, 0, -1):
eps = 1/(11-x)
db = DBSCAN(eps=eps, min_samples=10).fit(X)
core_samples_mask = np.zeros_like(db.labels_, dtype=bool)
core_samples_mask[db.core_sample_indices_] = True
labels = db.labels_
print(eps)
ax = fig.add_subplot(2, 5, i)
ax.text(1, 4, "eps = {}".format(round(eps, 1)), fontsize=25, ha="center")
sns.scatterplot(X[:,0], X[:,1], hue=["cluster-{}".format(x) for x in labels])
i += 1eps=0.1 と eps=0.3 の間にちょうどよいパラメーターがありそうですね。このあたりを微調整して、最適なパラメータを決めていくことになります。一例としてeps=0.18のときのコードと図を掲載します。
db = DBSCAN(eps=0.178, min_samples=10).fit(X)
labels = db.labels_
fig = plt.figure(figsize=(10, 10))
sns.scatterplot(X[:,0], X[:,1], hue=["cluster-{}".format(x) for x in labels])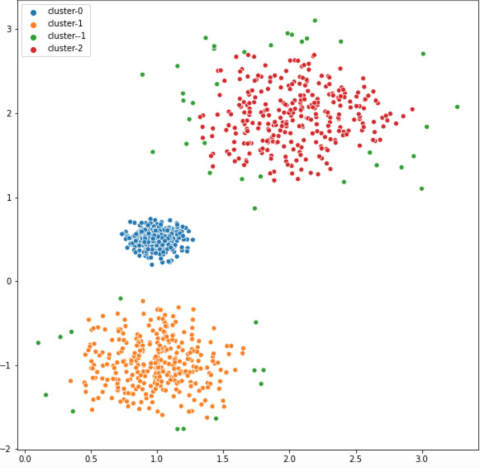
DBSCANの長所と短所
DBSCANにはk-meansなど他のクラスタリング法とは違ってクラスター数をあらかじめ決めなくていいという長所があります。
また、クラスターが再帰的に決定されていくので外れ値などのoutlierの影響を受けにくい性質があります。
逆に、計算コストは高いのでリアルタイム性が求められる場合には不向きですし、データが密集していると適切にεとminPtsを決めるのが難しいという欠点があります。
この記事は以上になりますが、DBSCANの仕組みをわかりやすく解説した動画があるのでさらに知りたい方は少し見てみてください。
関連図書
この記事に関連した内容を紹介している本はこちらです。
Google Colaboratoryでデータサイエンスを始めよう【使い方入門】
NumPyとPandasの意外と知らない10の関数 【コードあり】
今日も【生命医学をハックする】 (@biomedicalhacks) をお読みいただきありがとうございました。
当サイトの記事をもとに加筆した月2回のニュースレターも好評配信中ですので、よろしければこちらも合わせてどうぞ